TP 2
Mathematische großskalige Modellierung von Signalwegen in pankreatischen neuroendokrinen Tumoren (pNET)
Patienten-individuelle biologische Veränderungen (Mutation, Expression, Aktivierung) sollen in einem mathematischen MAPK/PI3K/mTOR-Modell abgebildet werden. Dieses Modell soll dann in der Lage sein, möglichst genaue Vorhersagen machen können, ob verfügbare oder zukünftig einzusetzende therapeutische Interventionen in diesen Signalwegen erfolgreich sind, oder ob mit „Escape-Mechanismen“ im Sinne einer Therapieresistenz zu rechnen ist. Hierzu werden verschiedene genetische Veränderungen und differentielle Expression (Gen/Protein) a) mit dynamischen MAPK/mTOR Signaturen und b) mit differentiellen Therapieantworten korreliert.
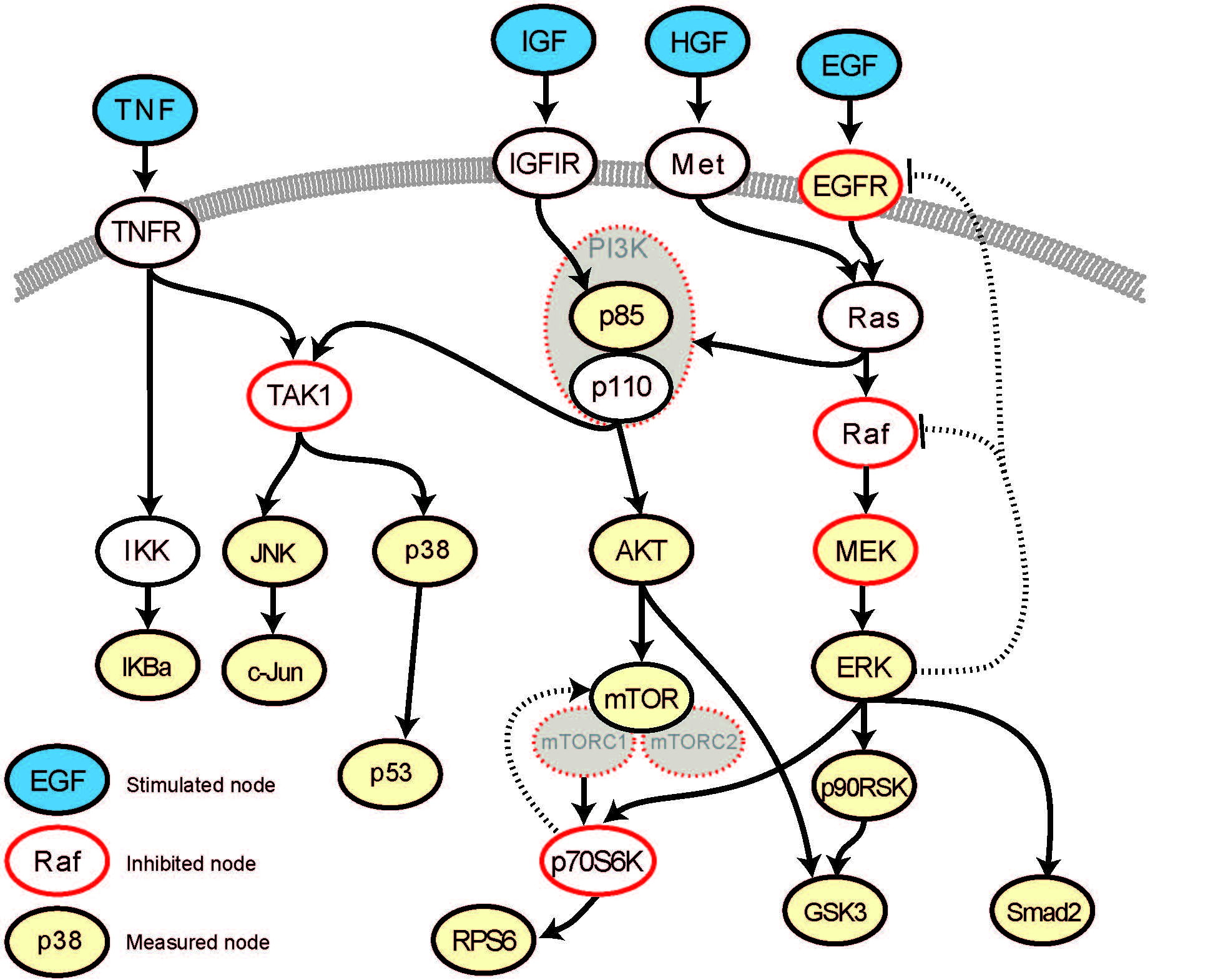
Die Hauptaufgabe des Teilprojekts der AG Blüthgen ist die Entwicklung und Simulation von patientenspezifischen Modellen der MAPK / PI3K / mTOR Signalwege. Technisch wird das Modell entwickelt werden durch (i), die Integration bestehender Modelle der MAPK / PI3K Signaltransduktion mit dem mTOR-Signalweg-Modell und Anpassung an neuroendokrine Tumorzellen, (ii) die Anpassung der Modellparameter unter Verwendung von Zeitreihen und Störungsdatensätzen, um Modelle zu erzeugen, welche die Signalübertragung bei Vorliegen bestimmter Mutationsmuster simulieren können, und (iii) den weiteren Ausbau der Modelle gegenüber wichtigen Knotenpunkten, die im Laufe des Projekts identifiziert werden.
Die Modelle werden zunächst mit einer in der AG Blüthgen etablierten Methode der Modular-Response-Analyse generiert, dann jedoch durch sogenannte Regressionsmodelle erweitert, um größere Netzwerkmodelle zu parametrisieren. Teilaspekte, bei denen die Zeit-Dynamik der Signalnetzwerke als wichtig erachtet werden, werden durch Differenzial-gleichungsmodelle abgebildet.
Darüber hinaus soll parallel zu den Signaltransduktionsnetzwerken die Auswirkungen der Signalnetzwerke auf die Phänotypen der Zellen (Proliferation, Migration, Zelltod) mit Hilfe von Regressionsmodellen oder anderen geeigneten statistischen Modellen modelliert werden, um die klinisch relevanten Prozesse vorhersagen zu können.
Außerdem übernimmt Nils Blüthgen die statistische Datenanalyse und entwickelt ein Model-assistiertes experimentelles Design, um die Experimente in den anderen Teilprojekten (AG Sers und AG Detjen) zu optimieren und somit die Modell-Experiment-Zyklen zu optimieren. Das Modell wird in der Lage sein, Modellvorhersagen zu generieren, die dann in verschiedenen experimentellen Modellen verifiziert werden können.
Keywords: mathematisches MAPK/PI3K/mTOR-Modell, Modular-Response-Analyse, Differenzialgleichungsmodelle



