TP 2
Erweiterung des SYSACT-Models basierend auf Boolescher Netzwerkanalyse und die Einbindung von TRAIL und MEK Signalnetzwerken
Das maligne Melanom ist die aggressivste Form des schwarzen Hautkrebses. Obwohl bemerkenswerte wissenschaftliche und klinische Erfolge zu verzeichnen waren, stieg die Anzahl der Neuerkrankungen in den letzten Jahrzehnten stark an. Häufig tritt die Mutation der BRAF-Kinase auf, die mit der Bildung von Metastasen korreliert. Dadurch entsteht eine besonders aggressive Form des Melanoms, die nur 20% der Patienten länger als 5 Jahre überleben lassen. Derzeit gibt es eine Chemotherapie, die spezifisch in den mutierten Signalweg eingreift und die Krebszellen abtötet. Leider werden die Krebszellen sehr schnell resistent und dies führt zu einer wiederkehrenden Ausbreitung des Krebses.
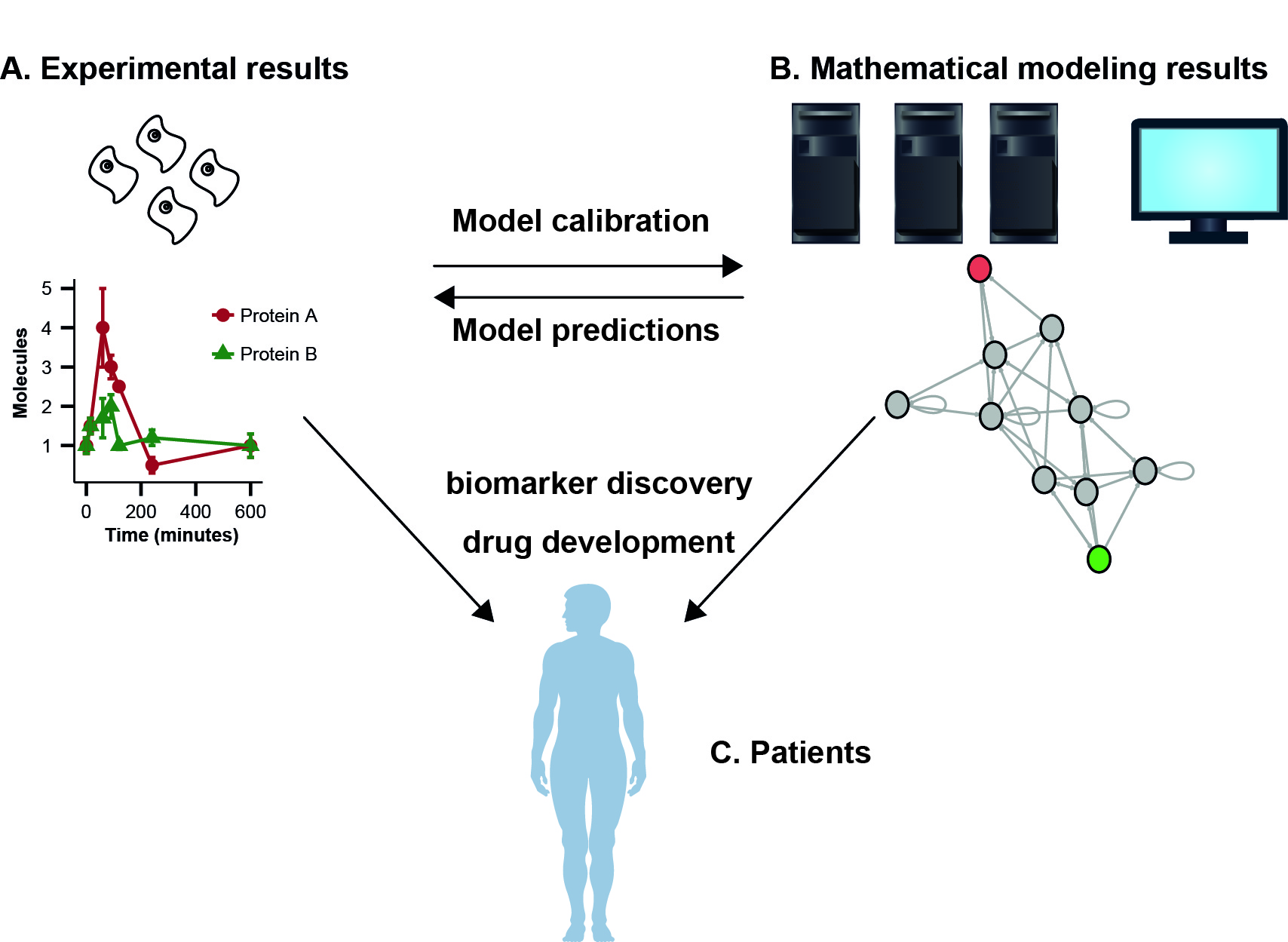
Wir werden mit mathematischen Methoden untersuchen, wie der programmierte Zelltod in unterschiedlichen Melanom-Krebszell-Linien spezifisch aktiviert werden kann. Für eine tiefgehende Analyse werden die mathematischen Modelle mit experimentellen Daten unterfüttert. Ein bereits veröffentlichtes Apoptose-Netzwerk (SYSACT-Modell, Passante et al., 2013, Cell Death Differ) dient hierbei als Referenz und soll auch hinsichtlich neuer experimenteller Daten untersucht werden. Aufgrund der große Datenmenge und der Größe des Netzwerkes bietet sich zur Analyse die Verwendung von probabilistischen Booleschen Netzwerken an (Trairatphisan et al., 2014, PLoS One). Diese Methode erlaubt die Modellierung der wahrscheinlichsten Proteininteraktionen und Regulationsstrukturen und ermöglicht die Anpassung der Aktivität der Signalmoleküle an die experimentellen Daten. Zudem werden die empfindlichen Netzwerkpunkte unterschiedlicher Zelltypen mit Sensitivitätsanalysen analysiert. Hierbei werden die sensitiven Netzwerkstrukturen vom MEK-pathway sowohl auch vom SYSACT-Model werden zu einem komplexen Model kombiniert. Damit lässt sich dann der Einfluss unterschiedlicher Wirkstoffe auf die interne Signalverarbeitung untersuchen und Möglichkeiten zur Optimierung des abtötenden Effekts können erarbeitet werden.
Das Ziel der Forschung liegt in der Identifizierung der relevanten Mechanismen, die zur Resistenzbildung in den verschiedenen Melanoma-Zelllinien beitragen. Sensitivitätsanalysen helfen bei der Entwicklung neuer Hypothesen zur Netzwerkstruktur, die im Labor experimentell geprüft werden können. Dies geschieht in enger Zusammenarbeit mit dem Labor für experimentelle Dermatologie an der Technischen Universität Dresden und dem Zentrum für Hauttumoren an der Universitätsklinik in Tübingen.
Keywords: Melanoma, SYSACT, Systems Biology, Probabilistic Boolean Network



