ComorbSysMed
Untersuchung von Komorbiditäten in entzündlichen Hauterkrankungen mittels Methoden des Maschinellen Lernens und Omics-Technologien in einem system-medizinischen Ansatz
Vorhabenbeschreibung:
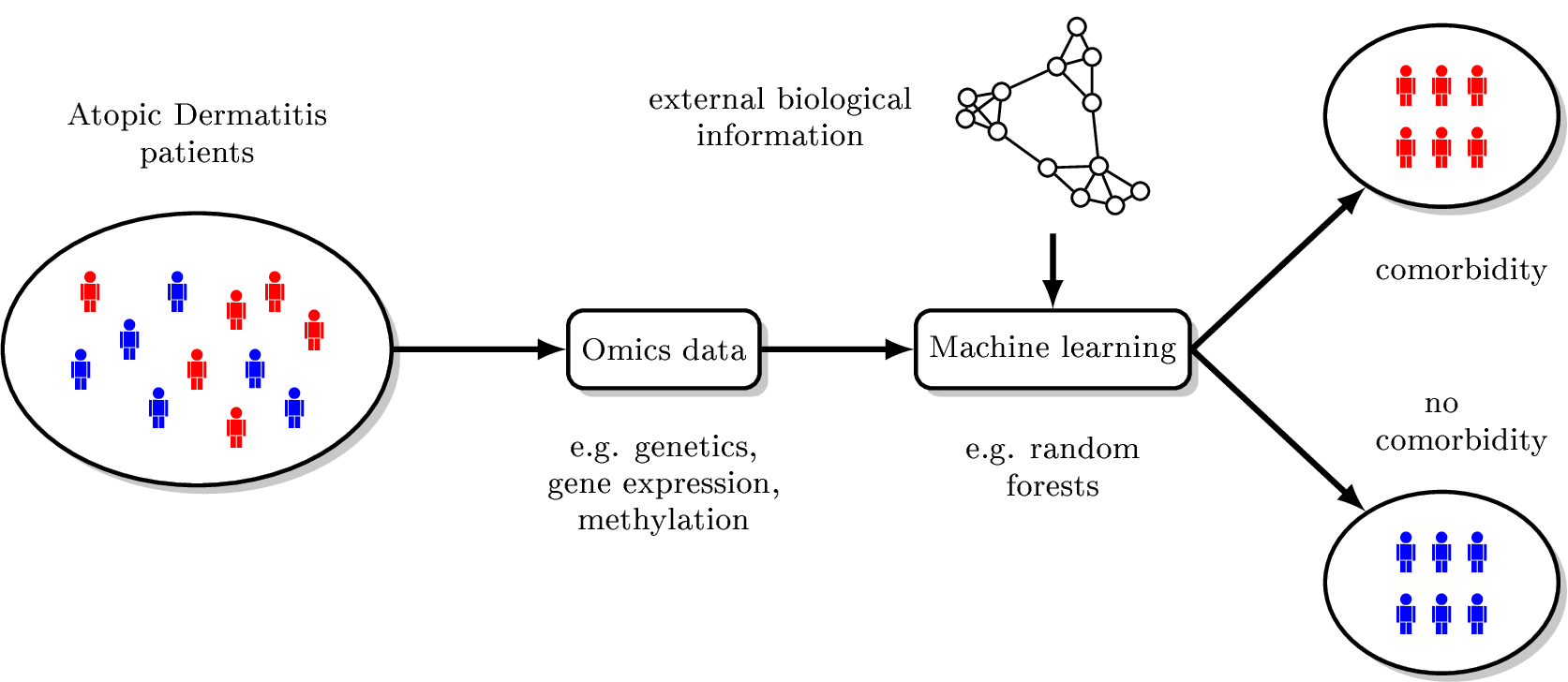
Patienten mit chronisch-entzündlichen Hauterkrankungen wie Neurodermitis und Psoriasis entwickeln häufig autoimmun-vermittelte Komorbiditäten. Um die zugrundeliegenden pathophysiologischen und molekularen Mechanismen zu verstehen, wird die Nachwuchsgruppe einen systemmedizinischen Ansatz verwenden, um verschiedene Omics-Daten sowie klinische Informationen und biologisches Wissen mithilfe von Verfahren des Maschinellen Lernens in mathematischen Modellen zu integrieren. Diese Modelle werden eine Stratifizierung der Patienten anhand ihres Komorbidität-Zustands sowie die Identifizierung von relevanten Molekülen, Pathways und Netzwerken ermöglichen.
Arbeitsplanung:
Ein Schwerpunkt der Nachwuchsgruppe ist die Entwicklung und Bewertung statistischer und informatischer Methoden, die eine Integration der verschiedenen Informationen ermöglichen. Zunächst geht es um Ansätze, mit denen aus der großen Menge an potentiellen Prädiktoren solche Variablen entfernt werden, die für die Klassifikation nicht relevant sind. Darüberhinaus werden Methoden untersucht, die strukturelles und funktionelles biologisches Wissen im Modellbildungsprozess berücksichtigen und unterschiedliche Arten von Omics-Daten integrativ analysieren. Um die Stärken und Schwächen der verschiedenen Ansätze vergleichen zu können, sind umfangreiche Simulationsstudien nötig, für die eine entsprechende Software- Struktur entwickelt wird. Zusätzlich werden die neu entwickelten statistischen und bioinformatischen Methoden als Pakete für die frei verfügbare und weit verbreitete statistische Software R implementiert werden.
Nach dem erfolgreichen Abschluss des methodischen Teils werden die identifizierten optimalen Methoden verwendet, um mathematische Modelle für Komorbiditäten bei Neurodermitis- und Psoriasis-Patienten zu entwickeln. Hierfür stehen verschiedene Patientenkollektive mit unterschiedlichen Omics-Messungen in Blut und Haut durch den klinischen Kooperationspartner zur Verfügung. Zusätzlich werden im Rahmen der Nachwuchsgruppe weitere Patienten rekrutiert werden, um die entwickelten Modelle in unabhängigen Proben zu validieren.
Publications
Degenhardt, F., S. Seifert, and S. Szymczak (2017). "Evaluation of variable selection methods for random forests and omics data sets." Briefings in Bioinformatics. ComorbSysMed doi.org/10.1093/bib/bbx124.
Gundlach, S., O. Junge, L. Wienbrandt, M. Krawczak, and A. Caliebe (2019). "Comparison of Markov Chain Monte Carlo Software for the Evolutionary Analysis of Y-Chromosomal Microsatellite Data." Comput Struct Biotechnol J 17: 1082-1090. CoMorbSysMed doi.org/10.1016/j.csbj.2019.07.014.
Hu, J., and S. Szymczak (2023). "A review on longitudinal data analysis with random forest." Briefings Bioinf 24(2). CoMorbSysMed doi.org/10.1093/bib/bbad002.
Kachroo, P., S. Szymczak, F.-A. Heinsen, M. Forster, J. Bethune, G. Hemmrich-Stanisak, L. Baker, M. Schrappe, M. Stanulla, and A. Franke (2018). "NGS-based methylation profiling differentiates TCF3-HLF and TCF3-PBX1 positive B-cell acute lymphoblastic leukemia." Epigenomics 10(2): 133-147. ComorbSysMed & SysINFLAME doi.org/10.2217/epi-2017-0080.
Schlicht, K., P. Nyczka, A. Caliebe, S. Freitag-Wolf, A. Claringbould, L. Franke, U. Vosa, B. Consortium, S. L. R. Kardia, J. A. Smith, W. Zhao, C. Gieger, A. Peters, H. Prokisch, K. Strauch, K. S. Group, H. Baurecht, S. Weidinger, P. Rosenstiel, M. T. Hutt, C. Knecht, S. Szymczak, and M. Krawczak (2019). "The metabolic network coherence of human transcriptomes is associated with genetic variation at the cadherin 18 locus." Hum Genet. SysINFLAME & ComorbSysMed www.ncbi.nlm.nih.gov/pubmed/30852652.
Seifert, S. (2020). "Application of random forest based approaches to surface-enhanced Raman scattering data." Sci Rep 10(5436): 1–11. CoMorbSysMed doi.org/10.1038/s41598-020-62338-8.
Seifert, S., S. Gundlach, O. Junge, and S. Szymczak (2020). "Integrating biological knowledge and gene expression data using pathway guided random forests: A benchmarking study." Bioinformatics. ComorbSysMed & coNfirm https://doi.org/10.1093/bioinformatics/btaa483.
Seifert, S., S. Gundlach, and S. Szymczak (2019). "Surrogate minimal depth as an importance measure for variables in random forests." Bioinformatics 35(19): 3663-3671. ComorbSysMed doi.org/10.1093/bioinformatics/btz149.
Tsoi, L. C., E. Rodriguez, F. Degenhardt, H. Baurecht, U. Wehkamp, N. Volks, S. Szymczak, W. R. Swindell, M. K. Sarkar, K. Raja, S. Shao, M. Patrick, Y. Gao, R. Uppala, B. E. Perez White, S. Getsios, P. W. Harms, E. Maverakis, J. T. Elder, A. Franke, J. E. Gudjonsson, and S. Weidinger (2019). "Atopic dermatitis is an IL-13 dominant disease with greater molecular heterogeneity compared to psoriasis." J Invest Dermatol. ComorbSysMed doi.org/10.1016/j.jid.2018.12.018.
Zivanovic, V., S. Seifert, D. Drescher, P. Schrade, S. Werner, P. Guttmann, G. P. Szekeres, S. Bachmann, G. Schneider, C. Arenz, and J. Kneipp (2019). "Optical Nanosensing of Lipid Accumulation due to Enzyme Inhibition in Live Cells." ACS Nano 13(8): 9363-9375. ComorbSysMed doi.org/10.1021/acsnano.9b04001.



