MultiPath
Ein generisches mehr-lagiges Modell für die Integration von Vorwissen aus unterschiedlichen Typen von Signalwegen
Vorhabenziele
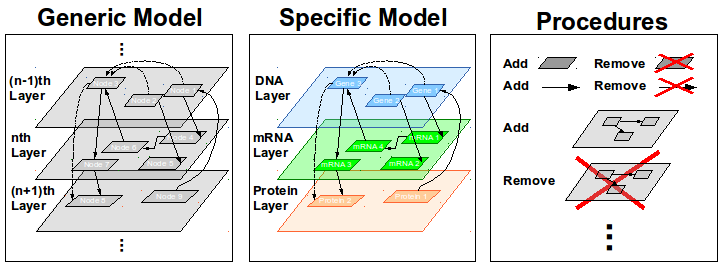
In der klinischen Forschung ist sowohl die Integration von Vorwissen über biologische Signalwege als auch das Nachvollziehen dieser Signalwege in wissenschaftlichen Publikationen ein sehr ineffizienter und umständlicher Vorgang. Das Ziel dieser Nachwuchsgruppe ist die Vereinfachung der Integration von Signalwegs-Wissen und die Stärkung von Reproducible Research in der klinischen Forschung im Allgemeinen und in der Systemmedizin im Speziellen. Die zentrale Idee ist hierbei die Definition und Umsetzung einer mehr-lagigen Modellierungsstruktur für biologische Signalnetze, welche ein generisches und erweiterbares Format für die Integration verschiedener Signalnetz-Typen und weiterer Quellen relevanten Wissens (z.B. Drug-Target-Datenbanken) bietet. Die besondere Eigenschaft dieser Modellierungsstruktur ist, dass sie Prozeduren für die automatische Transformation von Signalnetzen beinhaltet und diese reproduzierbar dokumentieren kann. Innerhalb dieser Nachwuchsgruppe sind zwei innovative Anwendungsfälle geplant:
- Erstens, die Unterstützung der klinischen Forschung durch das Erleichtern der Integration von Vorwissen zur Hypothesen-Generierung und für Analysen von Hochdurchsatz-Daten von Krebs-Kohorten.
- Zweitens, die Unterstützung der klinischen Praxis durch die Visualisierung von mehr-lagigen Signalnetz-Modellen mit integrierten Signalnetz-, Hochdurchsatz- und Mutations-Daten sowie Drug-Target-Datenbanken.
Arbeitsplanung
Das zentrale Ziel dieser Nachwuchsgruppe ist die Entwicklung einer mehr-lagigen Modellierungsstruktur für biologische Signalnetze. Dies umfasst die theoretische Ausarbeitung der Definition und die Implementierung des Modells für die praktische Anwendung. Neben einer eigenständigen Umsetzung der Software ist die Erweiterung von weit-verbreiteten Software-Tools mittels Plug-ins oder Software-Paketen zur Unterstützung dieser mehr-lagigen Modellierungsstruktur für biologische Signalnetze geplant. Hierdurch wird die Interoperabilität innerhalb der wissenschaftlichen Community verbessert und eine einfache Übernahme der Strukturen für andere Forscher ermöglicht. Zur Evaluierung dieser Signalwegs-Modelle werden vorhandene Wissens-Ressourcen in einem Online-Katalog von Modellen zusammengestellt. Weiterhin werden in Kooperation mit den klinischen Partnern spezifische Modelle an den Kohorten-Daten zu kolo-rektalen Karzinomen (DFG KFO179) und metastasierenden Tumoren (BMBF MetastaSys) erstellt und funktionell analysiert.
Keywords: Network Medicine, Network Biology
Publikationen
Auer, F., Z. Hammoud, A. Ishkin, D. Pratt, T. Ideker, F. Kramer and J. Kelso (2018). "ndexr-an R package to interface with the network data exchange." Bioinformatics (Oxford, England) 34(4): 716-717. MultiPath doi.org/10.1093/bioinformatics/btx683.
Auer, F., S. Mayer and F. Kramer (2022). "Data-dependent visualization of biological networks in the web-browser with NDExEdit." PLoS Comput Biol 18(6): e1010205. MultiPath doi.org/10.1371/journal.pcbi.1010205.
Hammoud, Z. (2021). Using n-layer graph models for representing and transforming knowledge on biological pathways. Dr. rer. nat., Georg-August-University Göttingen. https://ediss.uni-goettingen.de/handle/21.11130/00-1735-0000-0005-15A6-8
Hammoud, Z. and F. Kramer (2018). "mully: An R Package to Create, Modify and Visualize Multilayered Graphs." Genes 9(11). MultiPath doi.org/10.3390/genes9110519.
Hammoud, Z. and F. Kramer (2020). "Multilayer networks: aspects, implementations, and application in biomedicine." Big Data Anal 5(1): 1–18. MultiPath & coNfirm doi.org/10.1186/s41044-020-00046-0.
Hammoud, Z. and F. Kramer (2020). "Multipath: An R Package to Generate Integrated Reproducible Pathway Models." Biology 9(12): 483. Multipath & coNfirm doi.org/10.3390/biology9120483.
Kramer, F. and T. Beißbarth (2017). "Working with Ontologies." Methods in Molecular Biology (Clifton, N.J.) 1525: 123-135. MultiPath doi.org/10.1007/978-1-4939-6622-6_6.
Kramer, F., S. Just and T. Zeller (2018). "New perspectives: systems medicine in cardiovascular disease." BMC systems biology 12(1): 57. SYMBOL-HF, MultiPath & symAtrial doi.org/10.1186/s12918-018-0579-5.
Martinez-Hernandez, J. E., Z. Hammoud, A. M. de Sousa, F. Kramer, R. L. do Monte-Neto, V. Maracaja-Coutinho and A. J. M. Martin (2021). "Network-Based Approaches Reveal Potential Therapeutic Targets for Host-Directed Antileishmanial Therapy Driving Drug Repurposing." Microbiology Spectrum 9(2). MultiPath & coNfirm journals.asm.org/doi/10.1128/Spectrum.01018-21.
MultiPath-Projekte in den Medien:



