CLIOMMICS
Clinically-applicable, omics-based assessment of survival, side effects, and targets in multiple myeloma
Das Multiple Myelom (MM) ist eine maligne B-Zell-Erkrankung, die durch Vermehrung von klonalen Plasmazellen gekennzeichnet ist, welche im Knochenmark akkumulieren. Myelomzellen weisen eine große Anzahl an chromosomalen Aberrationen und Vielzahl an Änderungen in der Genexpression im Vergleich zu normalen Plasmazellen auf. Die Hauptmanifestationen des MM sind Knochendestruktionen, Anämie, vermehrte Infektionen und die Einschränkung der Nierenfunktion. Die mittlere Lebenserwartung der Patienten mit MM konnte in den letzten 20 Jahren auf 5 - 10 Jahre verlängert werden. Das MM ist bei den meisten Patienten immer noch nicht heilbar und damit lebensbegrenzend. Jedoch gelingt es, mehr komplette Remissionen (vollständige Rückbildung der Krankheit) zu induzieren, welche länger als 10 Jahre persistieren. Gleichzeitig werden durch neue Medikamente jedoch auch Nebenwirkungen, insbesondere Polyneuropathie (Erkrankungen des peripheren Nervensystems) und eine hämatologische (das Blut sowie die blutbildenden Organe betreffende) Toxizität, induziert.
Innerhalb des in vier eng verzahnte Bereiche (B1‐B4) aufgegliederten Verbundantrages „CLIOMMICS“ des Universitätsklinikums Heidelberg und des Deutschen Krebsforschungs-zentrums (DKFZ) Heidelberg werden umfassende Erkenntnisse der molekularen Charakterisierung (Interphase‐Fluoreszenz‐in‐situ‐Hybridisierung, Genexpressionsanalysen, RNA‐Sequenzierung, Analyse von Einzelnukleotid‐Polymorphismen) und der Bildgebung mit klinischen Daten zusammengeführt.
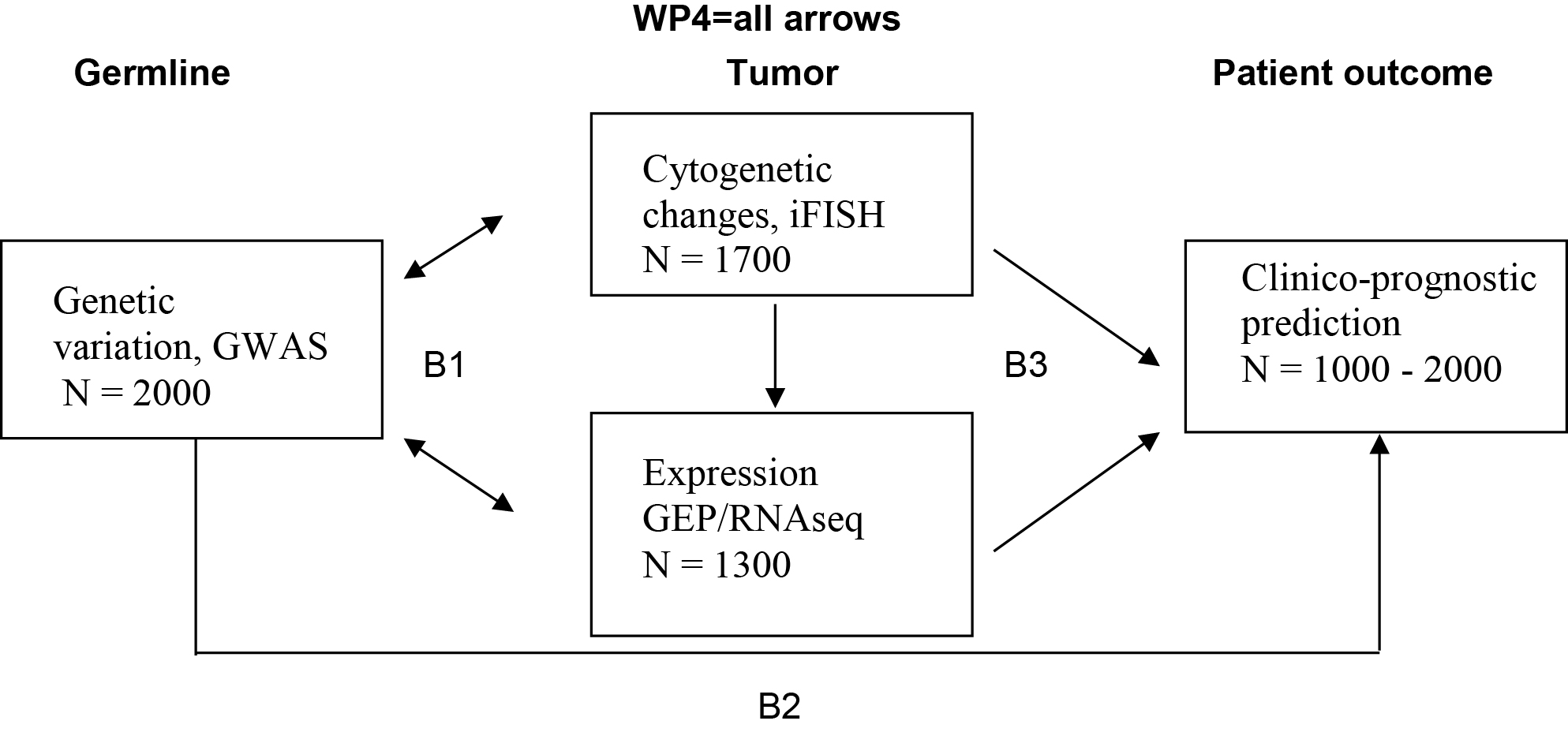
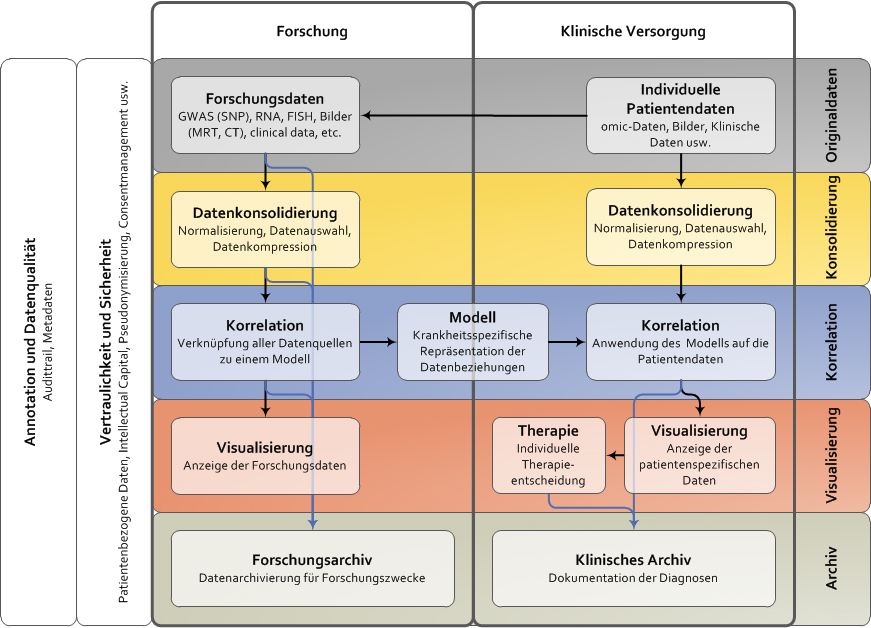
Für die bereits vorhandenen und in Teilprojekt B2 (genomweite Assoziationsstudien zur Prädiktion von Nebenwirkungen) sowie in Teilprojekt B3 erhobenen Daten (RNA‐Sequenzierung) werden in Teilprojekt B1 die IT-Infrastruktur und ein mehrstufiges Datenmanagementkonzept erarbeitet, das diese Daten integrieren kann. In Teilprojekt B4 werden potentielle und bekannte Prognosefaktoren aus allen Datenquellen kombiniert mit dem Ziel, ein integriertes statistisches Vorhersagemodell für den Krankheitsverlauf aufzustellen. Es wird ein „CLIOMMICS‐Report“ zur personalisierten Therapie von Patienten mit Multiplem Myelom erstellt, welcher eine bessere Prognoseabschätzung erlaubt und erstmals den Einsatz dieser Technologien in der klinischen Routine ermöglicht. Ziel ist die Verbesserung der Wirksamkeit der Myelomtherapie und die Prädiktion sowie Reduzierung von Nebenwirkungen.
Teilprojekte in CLIOMMICS:
TP 1 IT-Architektur und mehrstufiges Datenmanagement für Systemmedizin beim multiplen Myelom
TP 3 Transcriptomics by RNA-sequencing: Performing and reporting in clinical routine
Keywords: Multiple Myeloma, response to treatment, side effects, prognosis, gene expression, HM-metascore, integrative analysis, prediction of course of disease, patient report, multi level data management, IT architecture, systems medicine



