TP 2
Entwicklung und klinische Validierung neuer prognostischer Gewebemarker auf Basis von örtlichen und funktionalen Immunzellmustern
Ziel des Konsortiums ist es, entzündliche Reaktionen auf Tumorzellen bei erblichem Brustkrebs sowie auf Spenderorgane nach Nierentransplantation in Hinblick auf ihre prognostische Aussagekraft zu untersuchen und die gewonnenen Erkenntnisse klinisch zu validieren.
Im Rahmen des Teilprojekts von TP2 werden dazu Module zur robusten Bild- und Datenanlyse entwickelt und angewendet, die – aufbauend auf den Arbeiten aus der ersten Förderphase – den örtlichen Kontext von Immunzellverteilungen charakterisieren und dies nutzen, um Biomarker mit prognostischer oder prädiktiver Aussagekraft zur erzeugen. Darauf basierende vereinheitlichende Datenbanken werden erstellt und den Projektpartnern für ihre weiterführenden Arbeiten zur Verfügung gestellt.

In einem iterativen Prozess und in enger Zusammenarbeit mit den anderen Projektpartnern (insbesondere von TP4) wird interdisziplinär ein verbesserter, klinisch anwendbarer Workflow für dynamisch Interpretation von Immunzellmustern als prognostische und potentiell prädiktive Marker etabliert.
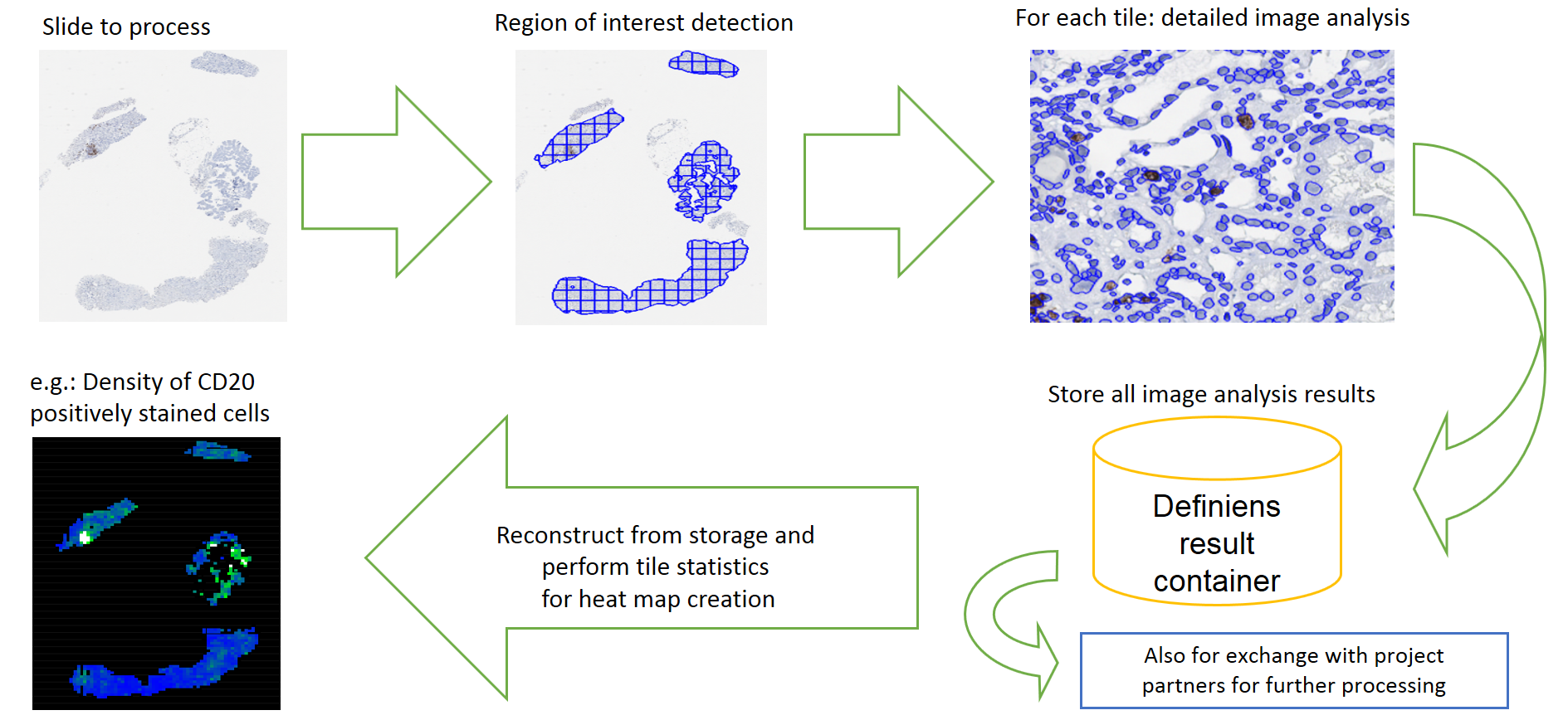
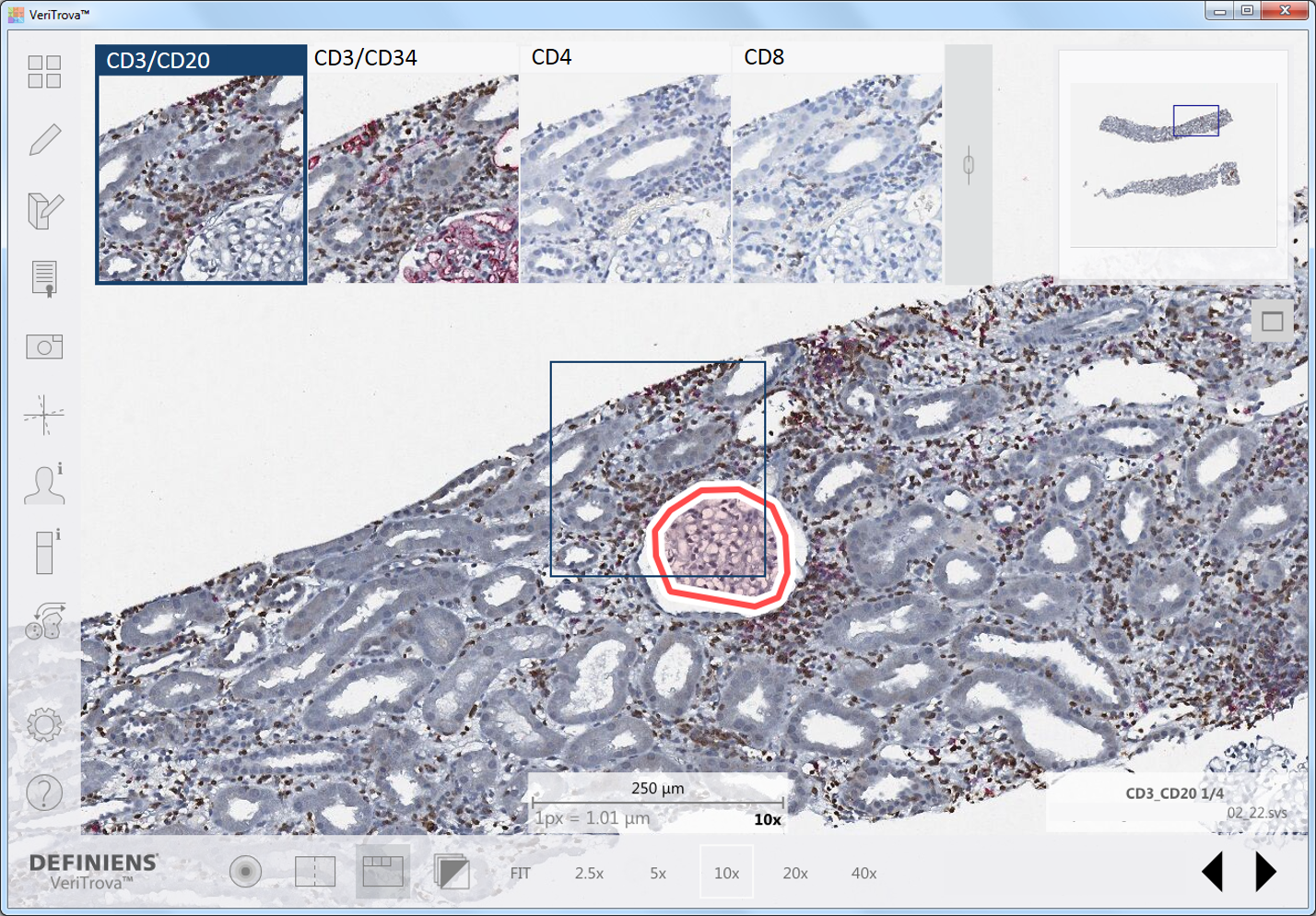
Bestehende Prototyp-Workflows zur Erfassung komplexer Beziehungen von Immunzellen im räumlichen Kontext von Brustkrebs und Transplantatabstoßung werden an die Erfordernisse klinischer Studien angepasst. Wie schon in der ersten Förderperiode besteht dabei für WP2 ein Schwerpunkt bei der Bereitstellung und Anwendung robuster Methoden der Bildanalyse, mit deren Hilfe vereinheitlichende Datenbanken erstellt werden, die die Grundlage für weitere Arbeiten bilden. Methoden des Data Mining werden weiterentwickelt, um klinisch relevante „Phenes“ zu entdecken und zu validieren. „Phenes“ sind dabei mathematische Beschreibungen von Orts- und Funktionsmustern aus Biopsien, die charakteristische Merkmale mit klinisch relevantem Wert für Prognose oder Prädiktion darstellen.
Keywords: Image, analysis, Tissue Diagnostics, Image Mining



