TP 2
Central Resource I: Genomics and Epigenomics
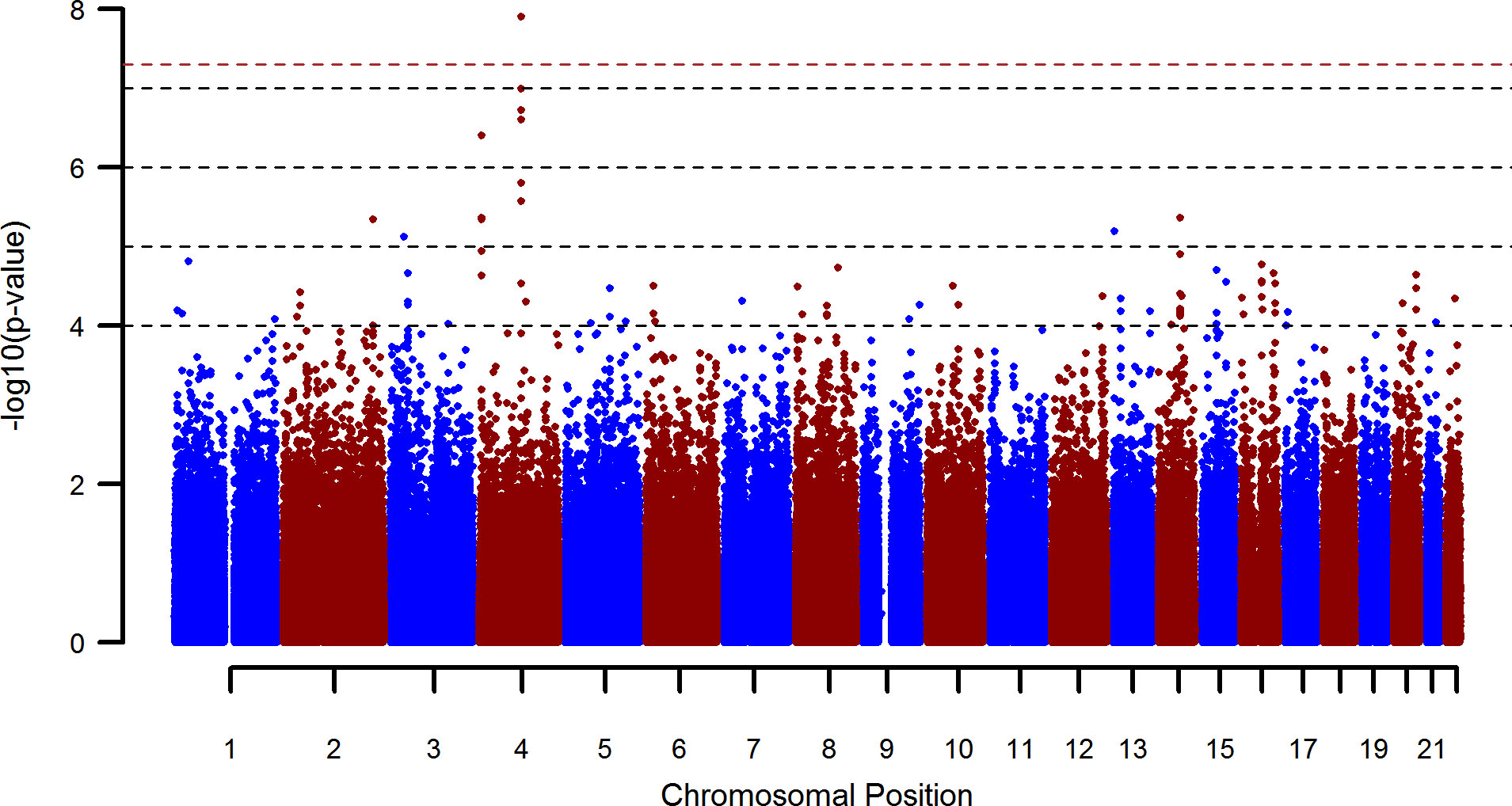
Alkoholabhängigkeit ist eine sehr verbreitete Erkrankung, die soziale und ökonomische Kosten zur Folge hat. Formalgenetische Studien haben gezeigt, dass die Erblichkeit der Alkoholabhängigkeit etwa 40–60% beträgt. In der Vergangenheit haben wir, gefördert vom Nationalen Genomforschungsprojekt, kandidatengenbasierte-, systematische genomweite- und Pathwayanalysen durchgeführt, auch unter Berücksichtigung von Daten aus Tiermodellen. Wir konnten mit diesem Ansatz weltweit erstmalig signifikante Befunde erzielen und die Validität des genetischen Ansatzes für ein Verständnis der biologischen Basis der Alkoholabhängigkeit zeigen (s. Abbildung). Diese Befunde wurden seitdem von den Ergebnissen anderer Arbeitsgruppen in unabhängigen Untersuchungen unterstützt. Durch weitere Untersuchungen konnten wir eine Korrelation eines dieser Gene (GATA4) mit Rückfallverhalten nachweisen, sowie dessen Einfluss auf die Funktion bestimmter Hirnareale. Zusätzlich belegten wir mit multi-marker-Analysen, dass heißt durch die gleichzeitige Untersuchung vieler Genvarianten, dass eine große Zahl weiterer Genvarianten mit jeweils kleinen Effekten zur Erkrankung beiträgt.
Das Ziel der Zentralen Ressource SP2 ist es diese genetischen Varianten und epigenetische Faktoren für Entstehung und Verlauf der Alkoholkrankheit zu identifizieren. Zu diesem Zweck wenden wir verschiedene Vorgehensweisen an: (i) Metaanalysen in internationalen Kollaborationen. (ii) Pathwayanalysen, um den gemeinsamen Beitrag von funktionell definierten Varianten mit kleinen Effektgrößen zu testen. (iii) Resequenzierung der meistversprechenden Gene um seltenere aber höherpenetrante Varianten zu identifizieren. (iv) Identifikation von Subgruppen von Patienten durch Untersuchung neuronaler Muster mittels bildgebender Verfahren (Endophänotypen), was die Eingrenzung auf homogenere Patientengruppen ermöglichen soll. (v) Analyse der Korrelation von genomweiter DNA-Methylierung mit unterschiedlichem Trink/und Abstinenzverhalten. Die umfassende Analyse von genomweiten Assoziationsdaten, epigenomischen Daten und Integration von diesen mit Befunden von anderen Subprojekten des Konsortiums sollen neue genetisch und epigenetisch regulierte Gene für Alkoholabhängigkeit identifizieren.



