TP 3
Central Resource II: Transcriptomics platform
Das Transkriptom ist der erste Punkt, an welchem die genetische Variabilität die Neurobiologie beeinflusst. Folglich können die angestrebten Ziele dieses Großprojektes nicht ohne umfangreiche Datenbanken über Genexpressionsunterschiede zwischen Alkoholikern und gesunden Probanden erreicht werden, da weder die Bedeutung genetischer Variationen noch die Bedeutung unterschiedlicher Gehirnaktivierungsmuster verstanden werden kann, ohne deren Einfluss auch auf molekularer Ebene zu analysieren. Die Aufgabe dieses Teilprojekts ist es deshalb, Transkriptionsdatenbanken von Gehirnen und induzierbaren pluripotenten Stammzellen (iPSC) und daraus differenzierten Neuronen von Alkoholikern und Kontrollpersonen sowie von Nagern relevanter Tiermodelle der Sucht zu erstellen.
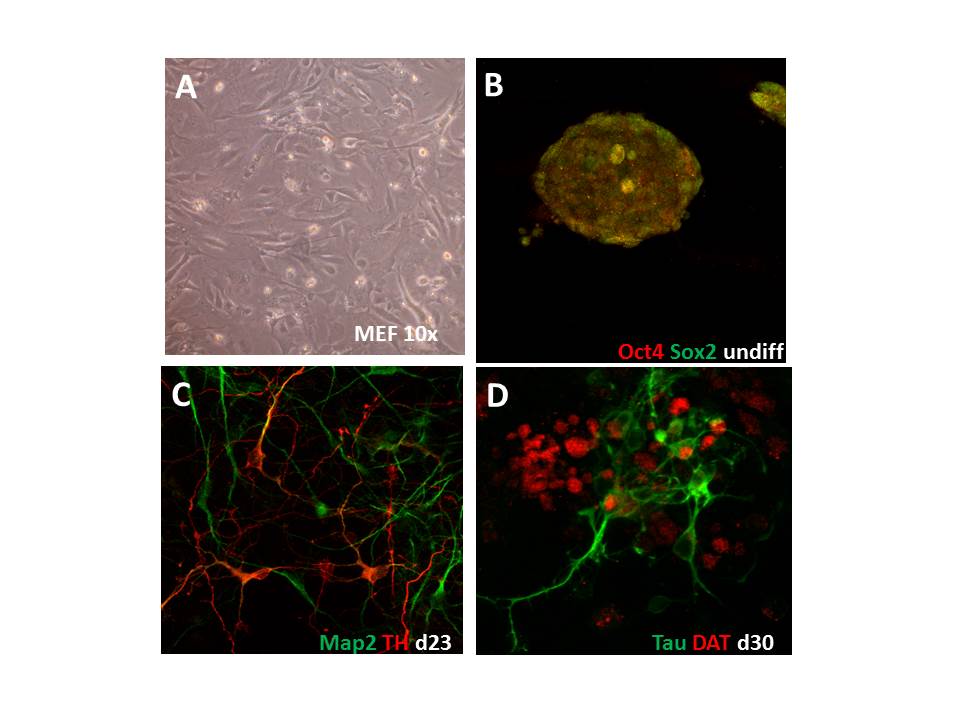
Diese Datenbanken werden die notwendige Information zur Verfügung stellen, um i) die in Teilprojekt SP2 gefundenen Einzelnukleotid-Polymorphismus (SNP) und DNA-Methylierungsstellen zu identifizieren und zu validieren; ii) potentielle Zielobjekte für eine funktionelle Validierung in den Teilprojekten SP9 und SP10 zu erkennen, und iii) eine ausführliche Liste der in den unterschiedlichen Experimenten der einzelnen Teilprojekte identifizierten Gene für die konvergierende Datenanalyse und Statistik in Teilprojekt SP6 zusammenzufassen. Zum einen werden wir humane Expressionsdaten belohnungsrelevanter Hirnregionen (z.B. präfrontaler Kortex, Striatum) aus postmortem Gehirnen sowie von Neuronen von Patienten mit hohem genetischem Suchtpotential, welche wir aus iPSC differenzieren, erstellen. Diese Proben sind die Grundlage, um die in Teilprojekt SP2 gefundenen SNPs auf mRNA und Proteinebene zu testen. Expressionsdaten von unterschiedlichen Tiermodellen des Alkoholismus sind schon aus früheren NGFN- und EU-geförderten Projekten zugänglich. Wir untersuchen konvergierende mRNA und microRNA Profile in humanen und murinen Gehirnpräparationen, wobei wir die RNA Profile in die genetischen Assoziations- und DNA-Methylierungstudien in Teilprojekt SP2 integrieren werden. Die Analyse der microRNA Profile soll einen Einblick in deren Bedeutung als Schlüsselposition bei der Regulation spezifischer Genexpression ermöglichen. Die kombinierten Daten werden die Grundlage für die bislang umfangreichste Datenbank des Alkoholismus und somit eine einzigartige Ressource sowohl für dieses Konsortium als auch für die wissenschaftliche Gemeinschaft sein.



