Quan-T-cell
Quantitative T-Zell-Immunologie zur Optimierung von Immuntherapie und Impfstrategien
Forschungsvorhaben:
Adoptive T-Zell-Therapie (ACT) zur Behandlung von Krebs und chronischen Virusinfektionen hat in jüngerer Vergangenheit zu vielversprechenden Therapieerfolgen geführt. Parallel dazu gab es immense Fortschritte bei der Therapie von fortgeschrittenen Tumorerkrankungen durch die pharmakologische Blockade von immunregulatorischen Checkpoints; mithilfe dieser Blockaden kann ein dysfunktionaler T-Zell-Zustand, "Erschöpfung" genannt, zum Teil aufgehoben werden. Trotz dieser Erfolge ist der Patienten-spezifische Nutzen dieser Immuntherapien durch heutige klinische Parameter nicht vorherzusagen und prospektive Biomarker zur Stratifizierung werden dringend benötigt. Während der Immunantwort differenzieren T-Zellen in verschiedene Effektor- und Gedächtnissubpopulationen mit unterschiedlichen Aufgaben. Die Abstammungsverhältnisse zwischen erschöpften T-Zellen und Gedächtnis- sowie Effektorsubpopulationen, die Plastizität dieser Subtypen und Möglichkeiten für molekulare Eingriffe sind allerdings immer noch schlecht untersucht. Wir haben vor kurzem gezeigt, dass datenbasierte mathematische Modellierung in Kombination mit der experimentellen Verfolgung von Einzelzellschicksalen eine deutliche Verbesserung bisheriger Zugänge zur Aufklärung von T-Zell-Differenzierungsprogrammen in vivo darstellt. In diesem kollaborativen Projekt nutzen wir diese Bündelung von mathematischer Modellierung und Experiment, um folgende Ziele zu erreichen:
- (1) Aufklärung der Mechanismen, die der T-Zell-Differenzierung und -Erschöpfung zugrunde liegen;
- (2) Die Anwendung dieser Ergebnisse zur Optimierung von ACT und pharmakologischer Therapie sowie von Impfstrategien.
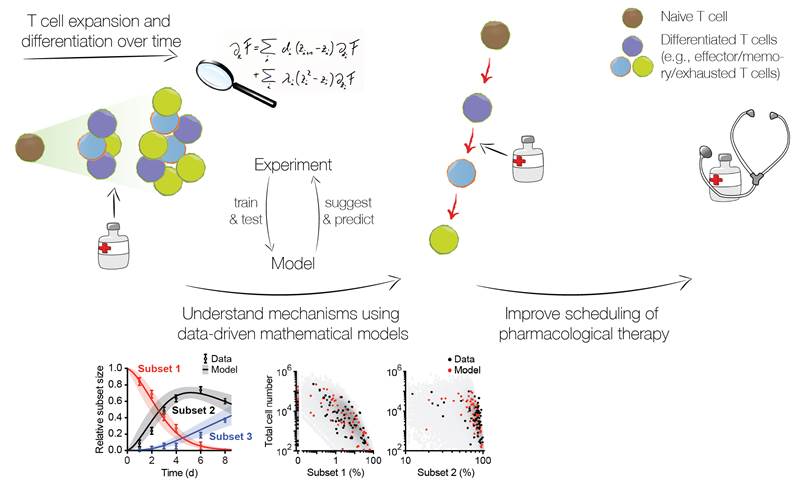
Arbeitsplanung:
Basierend auf präklinischen Mausmodellen zu chronischen Infektionen und dem Melanom, werden wir mathematische Modelle der T-Zell-Gedächtnisbildung und Erschöpfung entwickeln, um den bislang unbekannten dynamischen Zusammenhang zwischen terminal erschöpften und therapeutisch wertvollen T-Zell-Subpopulationen aufzuklären. Wir werden Differenzierungsmodelle aus den Daten inferieren, den Effekt immuntherapeutischer Medikamente quantifizieren, die zugrunde liegenden molekularen Netzwerke identifizieren und die Rolle der T-Zell-Avidität als potenziellen Parameter zur Vorhersage von T-Zell-Schicksal und therapeutischem Potenzial bestimmen. Diese mechanistischen Erkenntnisse werden wir dazu benutzen, die optimalen T-Zell-Subpopulationen für ACT zu identifizieren und die Planung pharmakologischer Therapien zu verbessern.
Publikationen
Cho, Y.-L., M. Flossdorf, L. Kretschmer, T. Höfer, D. H. Busch, and V. R. Buchholz (2017). "TCR Signal Quality Modulates Fate Decisions of Single CD4(+) T Cells in a Probabilistic Manner." Cell Reports 20(4): 806-818. Quan-T-Cell doi.org/10.1016/j.celrep.2017.07.005.
Flommersfeld, S., J. P. Bottcher, J. Ersching, M. Flossdorf, P. Meiser, L. O. Pachmayr, J. Leube, I. Hensel, S. Jarosch, Q. Zhang, M. Z. Chaudhry, I. Andrae, M. Schiemann, D. H. Busch, L. Cicin-Sain, J. C. Sun, G. Gasteiger, G. D. Victora, T. Hofer, V. R. Buchholz, and S. Grassmann (2021). "Fate mapping of single NK cells identifies a type 1 innate lymphoid-like lineage that bridges innate and adaptive recognition of viral infection." Immunity 54(10): 2288-2304 e2287. Quan-T-cell www.ncbi.nlm.nih.gov/pubmed/34437840.
Grassmann, S., L. Mihatsch, J. Mir, A. Kazeroonian, R. Rahimi, S. Flommersfeld, K. Schober, I. Hensel, J. Leube, L. O. Pachmayr, L. Kretschmer, Q. Zhang, A. Jolly, M. Z. Chaudhry, M. Schiemann, L. Cicin-Sain, T. Höfer, D. H. Busch, M. Flossdorf, and V. R. Buchholz (2020). "Early emergence of T central memory precursors programs clonal dominance during chronic viral infection." Nat Immunol 21(12): 1563–1573. Quan-T-cell doi.org/10.1038/s41590-020-00807-y.
Kretschmer, L., M. Flossdorf, J. Mir, Y.-L. Cho, M. Plambeck, I. Treise, A. Toska, S. Heinzel, M. Schiemann, D. H. Busch, and V. R. Buchholz (2020). "Differential expansion of T central memory precursor and effector subsets is regulated by division speed." Nat. Commun. 11(113): 1–12. Quan-T-Cell doi.org/10.1038/s41467-019-13788-w.
Pinto-Sietsma, S.-J., M. Flossdorf, V. R. Buchholz, J. Offerhaus, H. Bleijendaal, M. Beudel, P. G. A. Volders, R. M. A. ter Bekke, T. Dormans, P.-P. Zwetsloot, P. de Jager, S. Massberg, P. Rämer, C. Wendtner, E. Hoffmann, K. Rothe, S. Feihl, T. Kessler, Y. M. Pinto, and H. Schunkert (2020). "Antihypertensive drugs in COVID-19 infection." Eur Heart J Cardiovasc Pharmacother. AbCD-Net & Quan-T-cell doi.org/10.1093/ehjcvp/pvaa058.
Plambeck, M., A. Kazeroonian, D. Loeffler, L. Kretschmer, C. Salinno, T. Schroeder, D. H. Busch, M. Flossdorf, and V. R. Buchholz (2022). "Heritable changes in division speed accompany the diversification of single T cell fate." Proc Natl Acad Sci U S A 119(9). Quan-T-cell www.ncbi.nlm.nih.gov/pubmed/35217611.
Poschke, I., M. Flossdorf, and R. Offringa (2016). "Next-generation TCR sequencing - a tool to understand T-cell infiltration in human cancers." The Journal of Pathology 240(4): 384-386. Quan-T-cell doi.org/10.1002/path.4800.



