Sys_CARE
Systemmedizinische Untersuchung alternativer Spleißung bei Herz- und Nierenkrankheiten
Alternatives Spleißen (AS) ist ein biologischer Mechanismus durch den aus derselben DNA-Sequenz (Gen) verschiedene Transkripte (RNA-Kopien eines DNA-Abschnitts) und letztendlich verschiedene Proteine gebildet werden können.
Krankheitsbedingte Veränderungen von AS können die zelluläre Maschinerie auf mehreren molekularen Ebenen beeinflussen, wobei man davon ausgehen kann, dass der Einfluss einzelner Ereignisse gering ist. Der Einfluß von Änderungen des AS bei spezifischen Erkrankungen ist bisher nur unvollständig verstanden.
Um eine erste systemmedizinische Sicht auf AS zu etablieren, vereint Sys_CARE die Expertise biomedizinischer Forschung und klinischer Praxis (Universitätsmedizin Greifswald), sowie der Bioinformatik (Universität Hamburg und Helmholtz-Zentrum für Pharmazeutische Forschung Saarbrücken).
Sys_CARE wird dabei umfangreiche Multi-Omics Daten im Kontext von molekularen Interaktionsnetzwerken analysieren. Ein erster Schritt ist hierbei die Identifizierung von Transkripten die signifikant häufiger in Patienten zweier schwerer Erkrankungen, der dilatativen Kardiomyopathie (DCM), sowie der hypertensiven Nephropathie (HN), gefunden werden. Im Mittelpunkt der Analysen steht dabei der Einfluss von AS auf die Bildung von Proteinkomplexen mittels verschiedener Protein-Isoformen. Basierend darauf sagen wir Änderungen im Protein-Protein-Interaktionsnetzwerk vorher, die für die Krankheitsentwicklung von Bedeutung sein können. Dabei nutzen wir de novo Methoden zur Netzwerkanreicherung um Genexpressionsdaten mit Netzwerken zu integrieren und dadurch Mechanismen und regulatorische Programme zu entschlüsseln, die von AS betroffen sind.
Gleichzeitig werden wir die Rolle von AS in Verlaufsstudien an Maus- und Zebrafisch-Modellen der DCM und der HN untersuchen. Dadurch erhalten wir die Möglichkeit, Erkenntnisse über Organismen hinweg zu vergleichen. Ferner können wir so den zeitlichen Verlauf der Erkrankung untersuchen und ermitteln, inwiefern AS an der Krankheitsentstehung beteiligt ist.
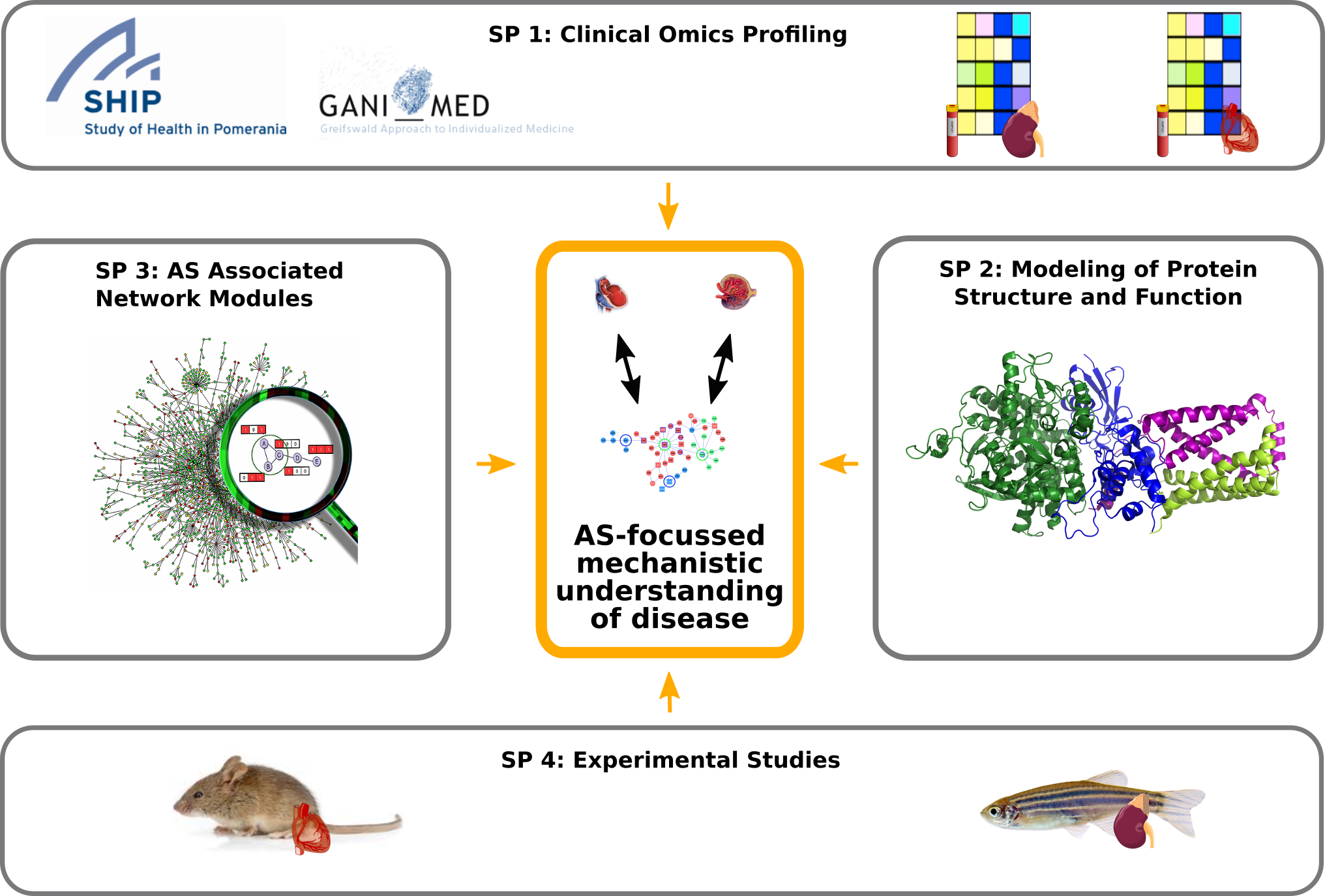

Projektorganisation in Sys_CARE: Das Zusammenwirken der vier Teilprojekte mit dem Ziel Netzwerkmodule zu identifizieren, die mit alternativem Spleißen in Verbindung stehen.
In silico Ergebnisse werden dabei in vitro und in vivo experimentell validiert. Deren Resultate wiederum dienen der iterativen Verbesserung unserer Netzwerkanalyse-Methoden. Wir erwarten, dass durch Sys_CARE gewonnene Einblicke in die Rolle von AS bei der DCM und der HN ein besonderes translationales Potenzial für die Entwicklung der Präzisionsmedizin mit sich bringen, da AS beispielsweise durch Antisense Oligonukleotide therapeutisch beeinflusst werden kann.
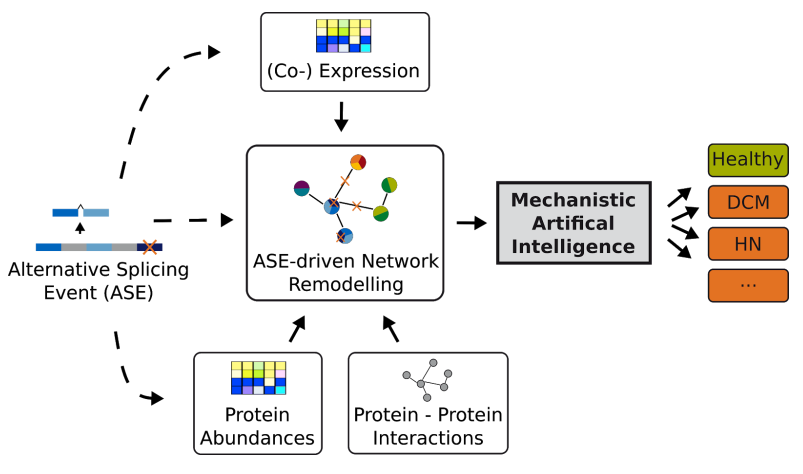
Das Sys_CARE Projekt zielt darauf ab unser mechanistisches Verständnis krankheitsrelevanter Formen des alternativen Spleißens zu verbessern. Dabei vergleichen wir Kohorten mit DCM und HN gegen eine Kohorte aus einer Bevölkerungsstudie (SHIP) um gemeinsame sowie krankheits-spezifische Ereignisse des alternativen Spleißens zu erkennen. Zusätzlich untersuchen wir Tiermodelle in Zeitverlaufsstudien um Einblicke in den Krankheitsverlauf zu erhalten.
Informationen zur Fördermaßnahme finden Sie auf der BMBF-Webseite



