SP 1 - DeepLTNBC
Identifizierung therapeutischer Biomarker durch Charakterisierung der Tumorheterogenität von tripelnegativen Brusttumoren auf Einzelzellebene
Eine möglichst umfassende Kenntnis der zellulären Zusammensetzung von Tumoren ist essentiell, um zu verstehen, wie sie entstehen und auf Therapien reagieren, und von höchster Wichtigkeit für die Entwicklung neuer Therapien. Bisherige Beschreibungen der Heterogenität von tripelnegativen Brusttumoren (TNBC) auf Einzelzellebene beschränken sich jedoch auf relativ wenige genetische Veränderungen oder untersuchen eine niedrige Anzahl von Patientinnen. Ausserdem ist die genetische Heterogenität nur ein Aspekt der Tumorvariabilität neben Unterschieden in Transkriptom, Proteom, epigenetischen Veränderungen und Morphologie.
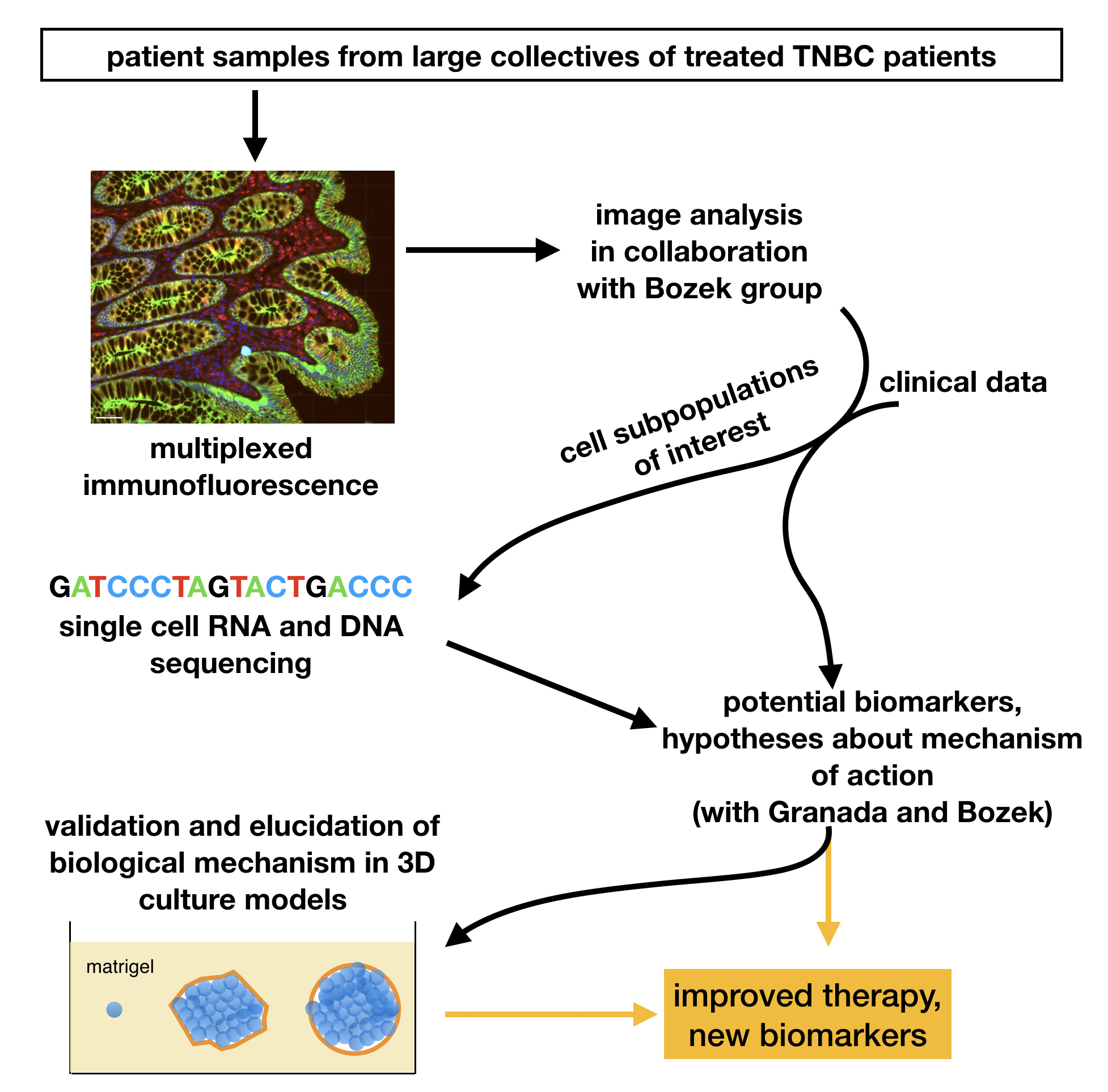
In diesem Teilprojekt werden wir multidimensionale Bild- und Sequenzdatensätze von großen Patientinnenkollektiven bis auf Einzelzellebene zu erzeugen ("-omics" Datensätze), welche die Heterogenität von tripelnegativen Brusttumoren vor und nach Therapie auf mehreren Ebenen um- fassend abbilden.
• Projektziel 1: Wir werden die Veränderungen in Menge und räumlicher Verteilung der Proteinex- pression von Tumorzellsubpopulationen unter Chemotherapie messen. Dazu werden wir neuar- tige "Multiplex"-Immunfluoreszenzverfahren verwenden, welche es erlauben, bis zu 60 Proteine auf einem einzigen Gewebeschnitt in subzellulärer Auflösung zu bestimmen.
• Projektziel 2: Wir werden Tumor-, Stroma- und Immunzellpopulationen aus dem Gewebe von Patientinnen mit TNBC aufreinigen und sie auf Genom- und Transkriptomebene charakterisieren.
Der integrierte Datensatz aus Projektziel 1 und 2 wird in Kollaboration mit den Gruppen Granada und Bozek mit HIlfe von künstlicher Intelligenz und mathematischer Modellierung analysiert werden, um Korrelationen zwischen den Veränderungen in der Tumorzell¬zusammensetzung und Prognose und Therapieerfolg zu finden.
• Projektziel 3: Wir werden Hypothesen entwickeln und validieren, wie gegen TNBC benutzte Medikamente das Tumorzellverhalten beeinflussen und zur Entstehung medikamentenresistenter Phänotypen führen. Dazu werden wir Experimente in 3D-Zellkulturmodellen für TNBC mit Ergebnissen aus den Projektzielen 1 und 2 kombinieren, um existierende Chemotherapieprotokolle zu verbessern und für jede einzelne Patientin individuell anzupassen.
Insgesamt werden wir in diesem Projekt neue Präzisionsmedizinstrategien entwickeln, die auf komplexen Kombinationen von Zelleigenschaften wie Proteingehalt und -lokalisation, Transkriptom, Genom, Morphologie und Zellumgebung beruhen.