SP 2 - DeepLTNBC
Deep Learning basierte Datenanalyse zur systemorientierten Charakterisierung von Triple Negative Breast Cancer
In diesem Projekt wir nutzen die neuen maschinellen Lernmethoden für Bilddatenanalyse, um neue Erkenntnisse über die Biologie von dreifach negativem Brustkrebs zu gewinnen, die bisher überwiegend mit Hilfe von DNA- und RNA-Sequenzierung untersucht wurden.
Bilddaten spielen in der Krebsforschung eine wichtige Rolle. Ob bei der Fluoreszenzmarkierung von Zellen oder in der Gewebemorphologie, reichhaltige biologische und diagnostische Informationen werden in Bildern kodiert. Im Gegensatz zu den digitalen Sequenzdaten ist die Extraktion und Quantifizierung relevanter Signaturen aus pixelcodierten Bilddaten jedoch eine anspruchsvolle Aufgabe. Der jüngste Aufstieg des Deep Learning hat neue Ansätze in der Bilddatenanalyse ermöglicht, indem er Algorithmen zur Quantifizierung von Informationen aus visuellen Daten durch maschinelles Lernen anbietet, ohne dass spezielle Verarbeitungsschritte entworfen werden müssen.
In Anwendung auf die Fragen zur TNBC-Biologie, die unser Projekt behandeln soll, in diesem Teil des Konsortiums wir entwickeln Ansätze zur Untersuchung von Bild- und Videodaten in 2D und 3D. Wir entwickeln überwachte Methoden, um in Bildern relevante, vom Menschen definierte Signale zu quantifizieren, wie z.B. Zellgröße oder Konzentration eines fluoreszierend markierten Moleküls. Wir einsetzen auch unbeaufsichtigte und teilüberwachte Modelle zum Erlernen der Bilddarstellung, die eine explorative Analyse der Bilddaten auf der Suche nach potenziell unbekannten visuellen Signaturen im Zusammenhang mit den Krankheitsmerkmalen und dem Krankheitsverlauf ermöglichen. Schließlich wir untersuchen die Zelldynamik in Krebsgewebe. Zusätzlich zur Bildanalyse wir durchführen alle bioinformatischen Sequenzanalysen und integrieren die verschiedenen experimentellen Modalitäten in eine umfassende systembiologische Datenanalysepipeline.
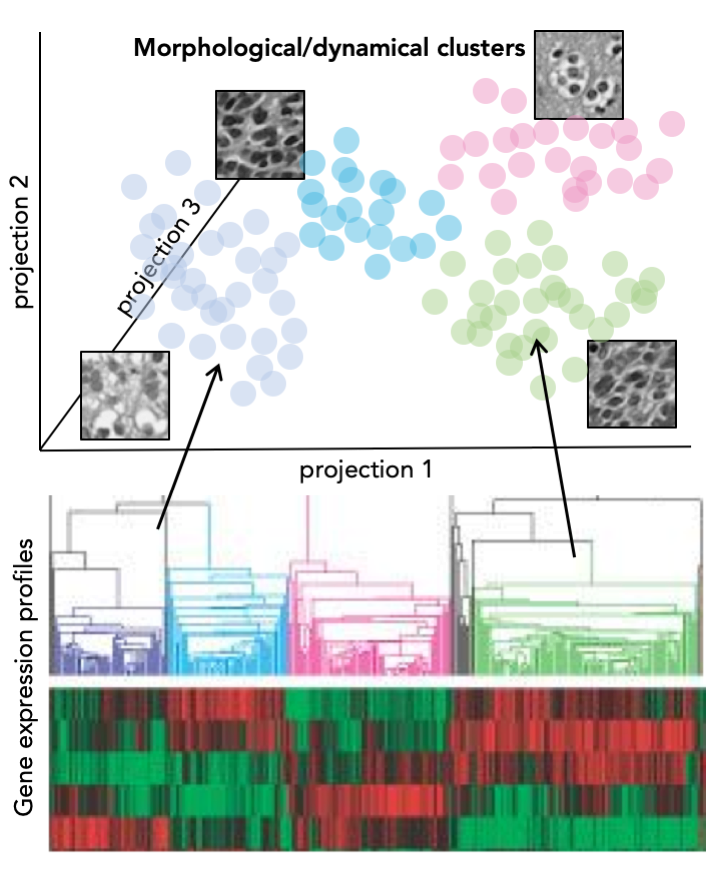

Wir entwickeln Methoden zum quantitatives Repräsentationslernen visueller Daten. Solche Repräsentationen ermöglichen eine systembiologische Analyse von Bildern in Kombination mit anderen molekularen Auslesungen mit hohem Durchsatz. Ziel dieses umfassenden Ansatzes ist es, neuartige, multimodale Biomarker für den sehr heterogenen dreifach negativen Brustkrebs zu finden.



