TP 3
Integrative Analyse zur Inferenz von kausalen Mutationen und Pathways einer Krankheit
Im vorliegenden Teilprojekt von MitOmics möchten wir die kausalen Mutationen und Pathways in Patienten mit mitochondrialen Störungen durch Integration von Genotyp, Transkriptomanalyse und genomweiten Pertubations-Versuchen an Zelllinien von betroffenen Patienten identifizieren.
Wir werden die Grundlagen und Algorithmen für folgende Arbeiten entwickeln:
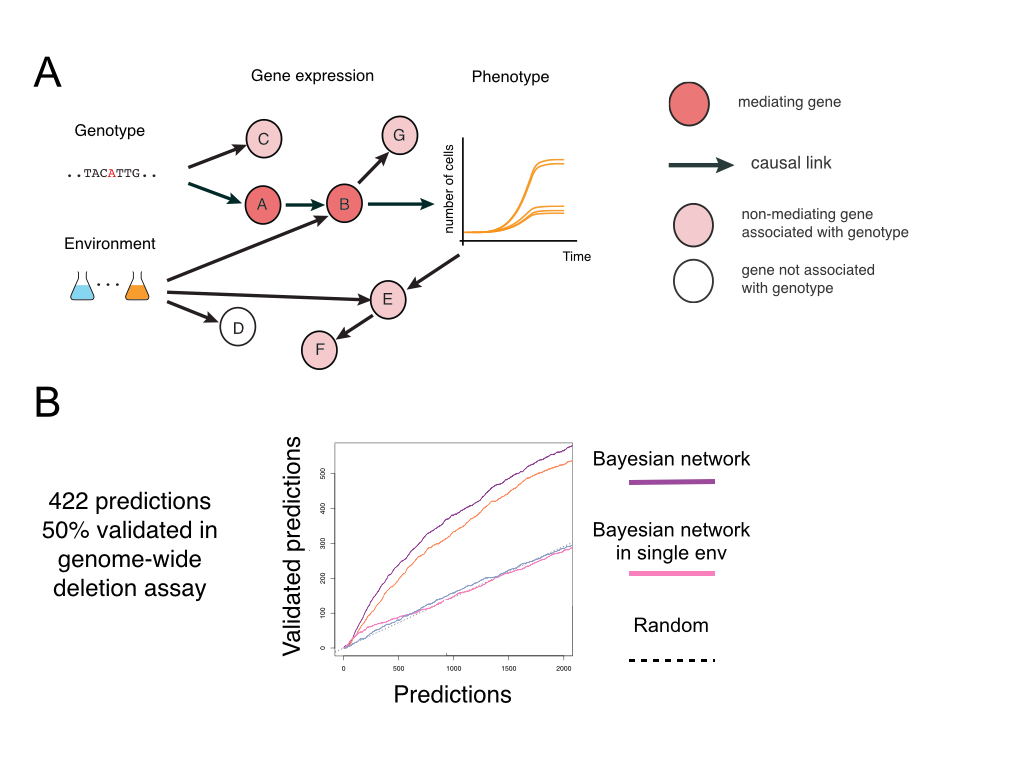
- (1) Identifizierung kausaler Mutationen und Pathways in individuellen Patienten. Wir möchten kausale Inferenzmethoden entwickeln, mit deren Hilfe wir kausale Mutationen und molekulare Ereignisketten (im Folgenden Pathways) aus Genotyp, Genexpression und phänotypischer Daten ableiten können. Die experimentelle Validierung unserer Vorhersagen erfolgt dann von unseren Kollaborationspartnern.
- (2) Statistische Analyse von pooled shRNA-Daten an Patienten-Zelllinien. Wir werden eine statistische Analysekette zur verlässlichen Bestimmung von umweltbedingten und Patienten-spezifischen Effekten eines pooled shRNA-Versuchs etablieren.