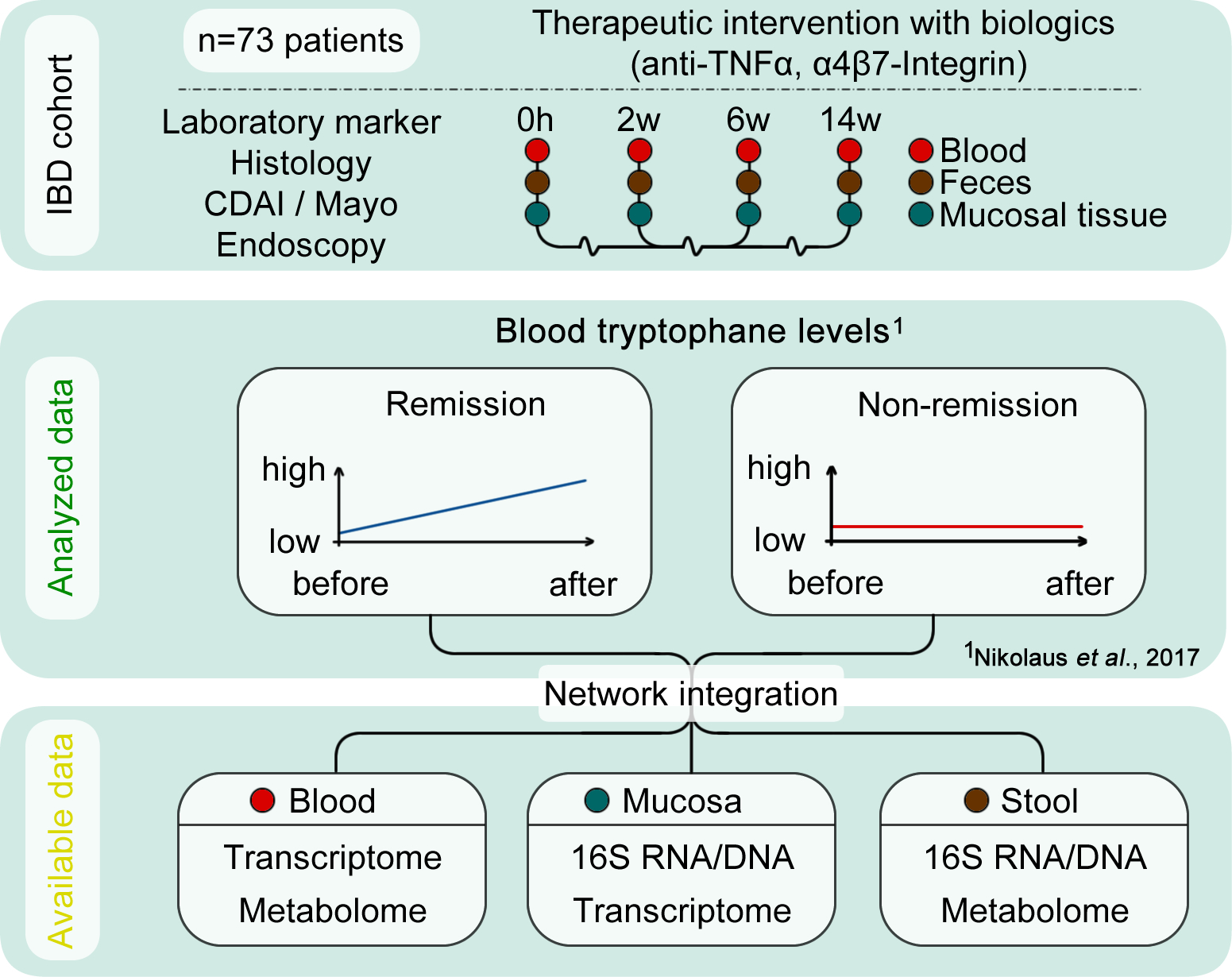
TP 2 - Try-IBD
Multi-Omics Datenintegration durch Graphen um Kompartment spezifische Molekuehle zu identifizieren welche den Tryptophan abhaengigen Immunmetabolismus in chronischen Darmerkrankungen beeinflussen.
Heutzutage besteht der gebräuchlichste Ansatz der Multi-Omics-Datenintegration darin, alle ‚Omic-Ebenen‘ unabhängig voneinander zu analysieren und anschließend nach gemeinsamen Merkmalen zu suchen. Bei dieser einfachen Strategie wird indirekt davon ausgegangen, dass alle Omics-Ebenen gleichermaßen zu einem biologischen Phänotyp beitragen, was zu einer Fehlinterpretation der Ergebnisse oder zum Übersehen relevanter synergistischer Effekte führen kann. Darüber hinaus erfasst diese schlichte Analyse nicht das komplexe Zusammenspiel „Cross-Omic“. Es gibt einige Anwendungen, in denen alle verfügbaren Omic-Ebenen zur Vorhersage klinischer Ergebnisse oder zur Durchführung einer Ontologieanalyse berücksichtigt werden. Leider erfordern diese meisten dieser Methoden mehrere hundert Proben, um das Zusammenspiel genau zu modellieren.
Hier am Institut of Compuational Biology, HelmoltzZentrum Muenchen, wurde ein auf Regression basierenden Network Inferenz-Ansatz entwickelt, der schon vorhandenes Vorwissen verwendet, um auf krankheitsspezifische mRNA / miRNA-Interaktionsnetzwerke zu schließen. Diese Methode wurde nun erweitert, um auf mehr als zwei Omic-Ebenen zu arbeiten und, übertrifft konkurrierende Techniken nicht nur hinsichtlich kleinen Kohorten (n = 50 Patienten), sondern auch bezüglich der Genauigkeit.
In TRY-IBD werden wir diese Methode verwenden fuer die (i) Netzwerk-Inferenz für Multiomic-Daten zur Identifizierung von Entwürfen für Schlüsselmerkmale. Darauf aufbauend wird eine (ii) Pfad zentrierte Analyse von Multiomics-Daten um wichtige IBD Zellfunktionen zu identifizieren und weiters wird eine (iii) Netzvalidierung und -erweiterung unter Verwendung von In-vitro-Co-Kulturmodellen erarbeitet. Letzten Endes werden die so erlangten Erkenntnisse verwendet um (iv) einen, auf Maschinellem lernen basierenden, Algorithmus zu erstellen welcher molekulare Subtypen von IBD klassifiziert die den therapeutischen Erfolg von IBD beeinflussen.