TP 3
Funktionelle Genomik der humanen Herzinsuffizienz
Dieses Projekts ist ein systembiologisch basierter Ansatz zur Identifikation molekularer Mechanismen, die für die Entwicklung der Herzinsuffizienz eine Rolle spielen. Hierfür steht ein großes Kollektiv von Herzinsuffizienzpatienten zur Verfügung. Durch extensive Charakterisierung des Genoms, des Transkriptoms und des miRNoms werden wir Signalnetzwerke identifizieren, die für die Pathogenese der Herzinsuffizienz eine wichtige Rolle spielen. Weiterhin stehen uns patientenspezifische Modelle zur Verfügung (Mausmodelle und iPS Zell-Linien, die humane Herzinsuffizienzmutationen imitieren), um die interessante Signaltransduktionskaskaden genauer zu evaluieren.
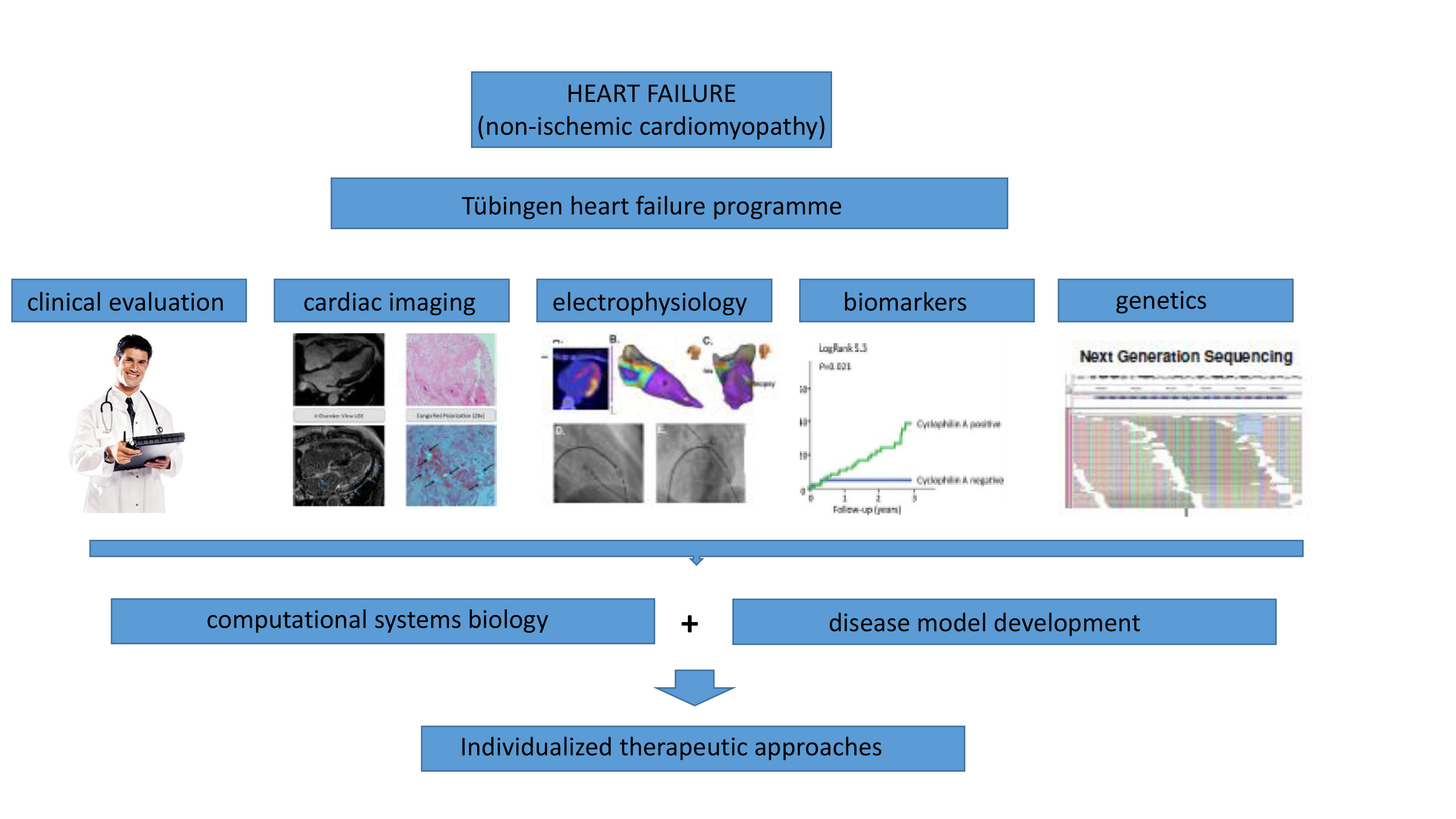

Am Herzkompetenzzentrum Tübingen werden Patienten mit Herzinsuffizienz systematisch aufgearbeitet. Dies beinhaltet neben der klinischen Charakterisierung eine ausführliche Bildgebung (inkl. Kardio-MRT, PET-CT), Herzkatheteruntersuchung, Herzbiopsie, Elektrophysiologie, Analyse von Biomarkern und Biosignalen sowie des Genoms. Die Daten werden in einem systembiologischen Ansatz ausgewertet, um Prädiktoren für schlechtes Outcome oder Herzrhythmisstörungen zu erarbeiten. Gleichzeitig entwickeln wir experimentelle Herzinsuffizienzmodelle (Maus- und Zellkulturmodelle), an denen neue, individualisierte Therapieformen getestet werden können.
Das Teilprojekt greift auf 3 Modelle zurück:
- 1. Charakterisierung von Patienten mit terminaler Herzinsuffizienz
Uns stehen über 180 Myokardproben von Patienten mit terminaler Herzinsuffizienz zur Verfügung. Diese Proben werden einem ausführlichen miRNA- und mRNA Profiling unterzogen und genotypisiert. So werden wir in der Lage sein, verschiedene Mutationen und mRNA/ microRNA-Profile mit Krankheitsverläufen zu assoziieren.
- 2. Charaktisierung einer Mauslinie mit Herzinsuffizienz (Titin knock-in Mauslinie)
Wir planen ein ausführliches miRNA-Profiling einer Herzinsuffizienz-Mauslinie zu verschiedenen Zeiten der Krankheitsentstehung. Interessante miRNAs können anschließend im Mausmodell durch Antagonisierung(„antiMIRs“) oder Überexpression („mimics) herausgearbeitet werden. Zellkulturexperimente werden uns helfen, wichtige miRNAs weiter zu charakterisieren
- 3. Charakterisierung von induzierten pluripotenten Stammzellen (iPS) Zellen von Patienten mit Herzinsuffizienz
Es wird ein mRNA/ miRNA Profil einer iPS Linie erstellt, welche eine Titinmutation trägt. Durch Überexpressions- und silencing Experimente wird die Bedeutung einzelner miRNAs herausgearbeitet werden. Weiterhin werden neue iPS Linien von Trägern anderer Mutationen erstellt und mit der Titin-Linie verglichen.
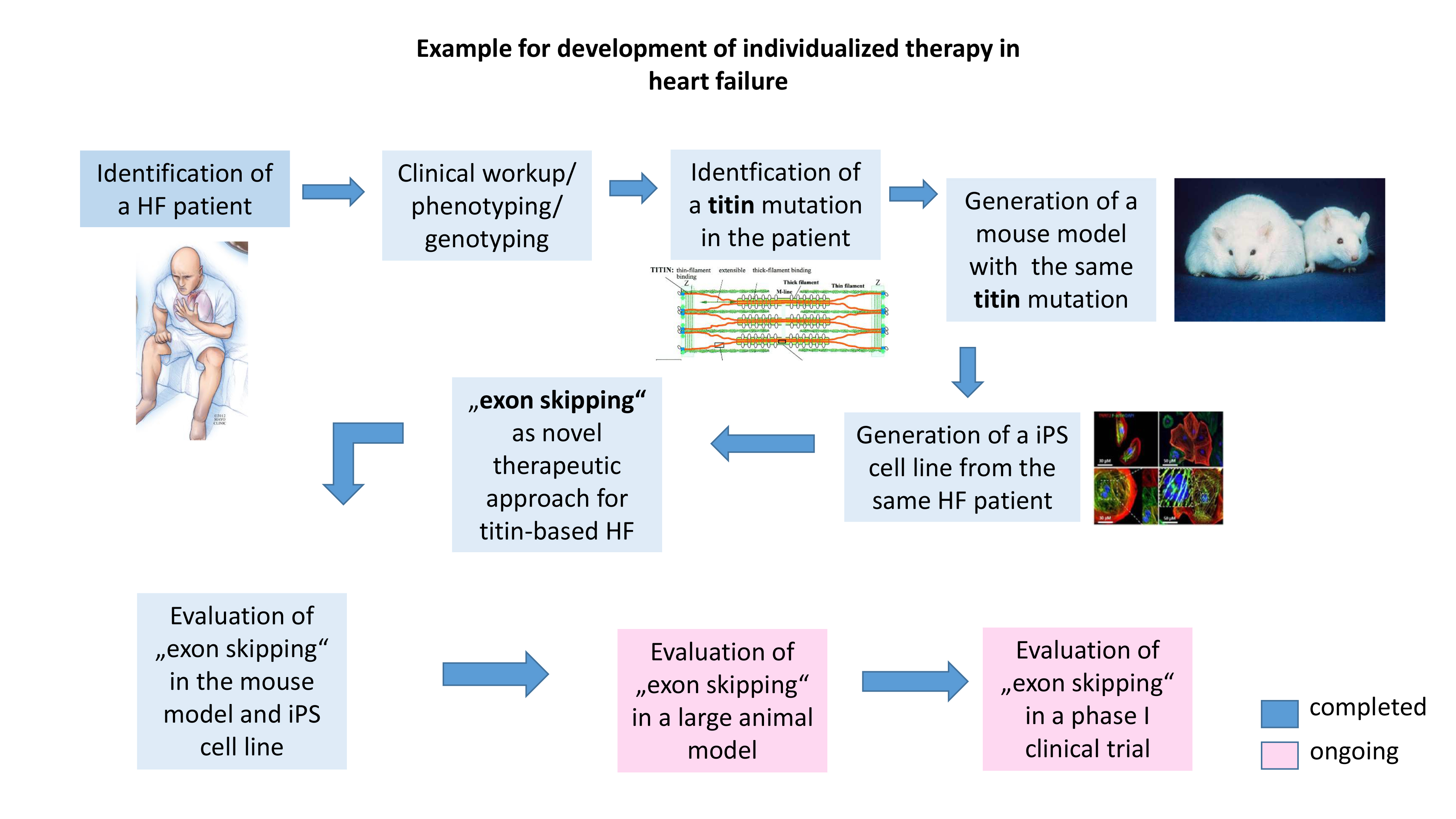

Die genetische Analyse eines Herzinsuffizienzpatienten erbrachte eine Mutation im sogenannten Titin als Ursache der Erkrankung. Wir haben experiementelle Krankheitsmodelle (Mausmodell und iPS-Zellkulturmodell) generiert, welche die extakt identische Titin-Mutation tragen wie der betroffene Patient. Anschließend haben wir einen experimentellen Therapieansatz entwickelt (sogennantes "exon-skipping") und dies an unseren Krankheitsmodellen erfolgreich getestet. Momentan evaluieren wir diese Therapie im Großtiermodell, bevor sie am betroffenen Patienten angewendet werden kann.



