TP 3
Dynamische Modellierung des Antwortverhaltens auf Wirkstoffe in HER2-low Brustkrebszellen
Die zentrale Fragestellung dieses Teilprojektes ist die Entschlüsselung und quantitative Beschreibung der Wirkmechanismen verschiedener therapeutischer Antikörper, die zur Behandlung von Brustkrebs des Typs HER2-low eingesetzt werden. Hierfür werden biologisch motivierte, mathematische Modelle der Zelle entworfen, um die Interaktionen der Rezeptoren und der relevanten Proteine von Signalwegen zu beschreiben.
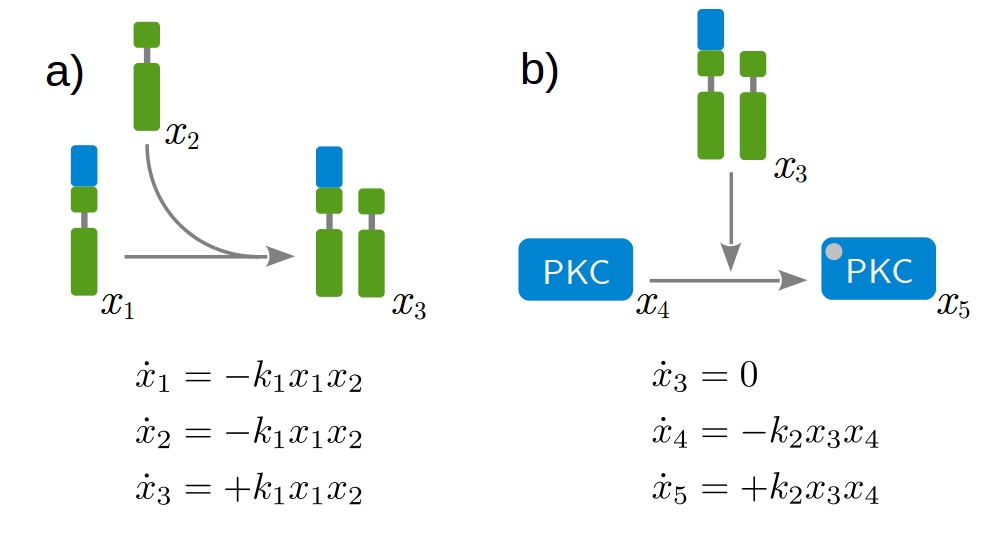
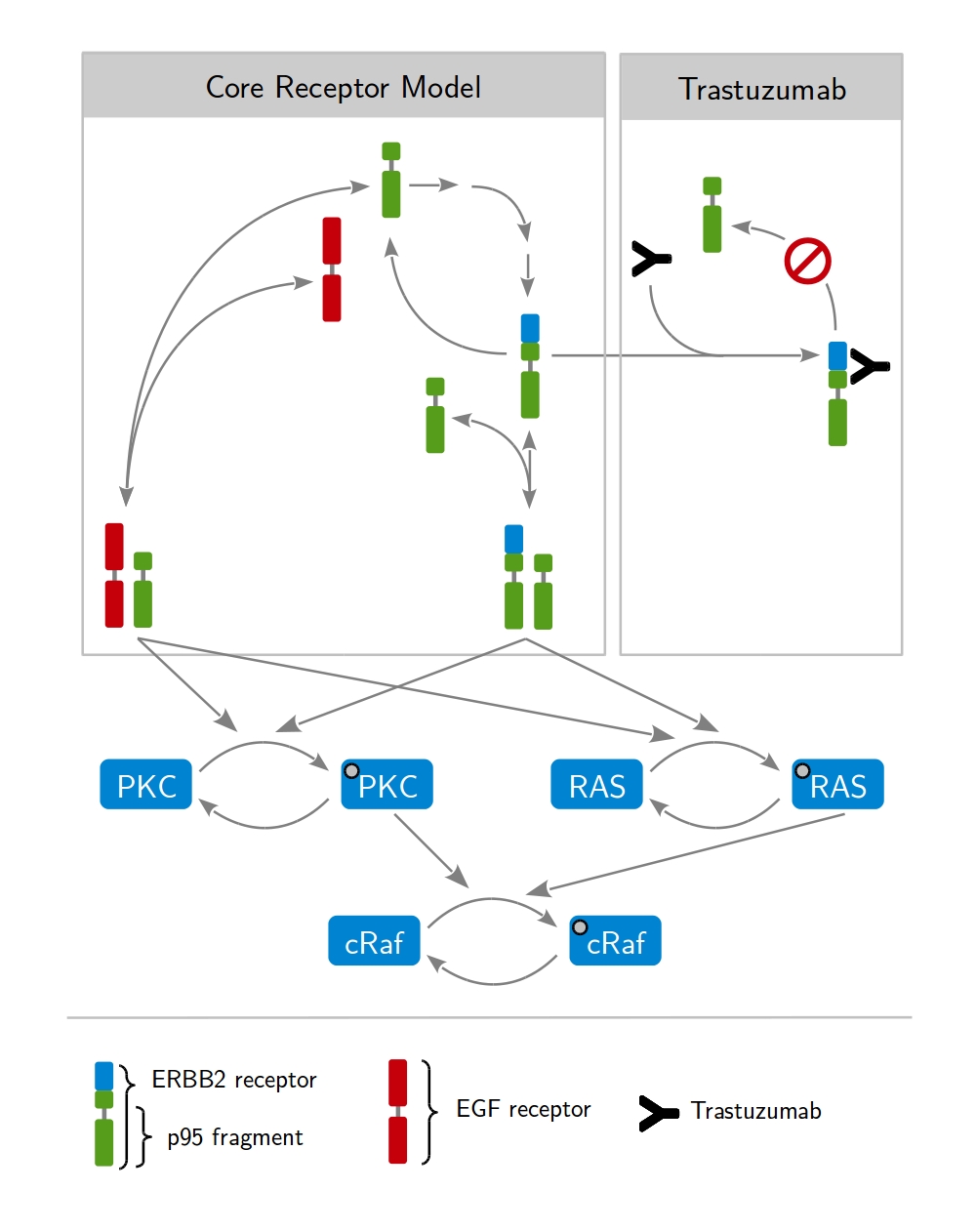
Die Methodik basiert auf dynamischer Modellierung. Darunter ist zu verstehen, dass die zeitliche Entwicklung des biologischen Systems durch Differentialgleichungen beschrieben wird. Diese drücken die Zu- oder Abnahme der modellierten Proteinkonzentrationen aus. Die Dimerisierung der Rezeptoren und die anschließende Phosphorylierung intrazellulärer Proteine sind hierbei grundlegende Prozesse, die schematisch in Abb. 1 zu sehen sind. Aus den verschiedenen möglichen Interaktionen ergibt sich damit ein großes Netzwerk gekoppelter Differentialgleichungen. Eine stets wichtige Frage dabei ist, welche Prozesse relevant für die gegebene Fragestellung sind und daher in das Modell mit aufgenommen werden. In Abb. 2 ist ein Modell zu sehen, das für die Beschreibung der Wirkungsweise von Trastuzumab in HER2-high Brustkrebszellen entwickelt wurde.
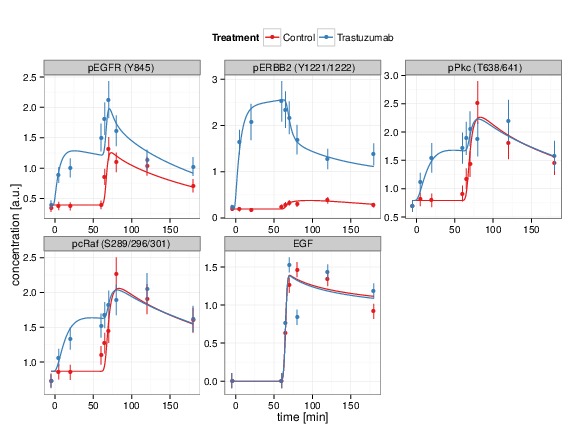
In den Interaktionstermen tauchen Reaktionsraten auf, die in der Regel unbekannt sind und aus experimentellen Daten bestimmt werden müssen. Als wesentliche Grundlage dienen zeitaufgelöste Messungen der Proteinkonzentrationen, die von den experimentellen Projektpartnern zur Verfügung gestellt werden. Um die Parameter zu bestimmen, welche eine optimale Beschreibung der Daten durch das Modell erlauben, wird eine Bewertungsfunktion aufgestellt, die sogenannte Log-Likelihood. Sie gibt die Übereinstimmung von Modell und Daten wieder, so dass die Parameterwerte am Maximum die passendste und somit plausibelste Konstellation darstellen. Aus dem Ergebnis der Parameteranpassung lässt sich auch erkennen, ob das Modell überhaupt die Daten ausreichend beschreiben kann, wie in Abb. 3 gezeigt, oder ob eine Anpassung des Modells erforderlich ist.
Im Rahmen dieses Projektes werden bereits etablierte dynamische Brustkrebs-Modelle weiterentwickelt, um den Wirkmechanismus von gezielten Wirkstoffen in unterschiedlichen Krebszelllinien zu untersuchen. Aus den gewonnenen Erkenntnissen sollen neue Strategien zur Optimierung der Medikamentenwirksamkeit entwickelt werden.