TP3
Computer-gestützte Modellierung der mitochondrialen Dysfunktion
Dieses Teilprojekt wird biomolekulare und genomische Datensätze für Parkinson-Patienten und Kontrollpersonen (aus den ersten beiden Teilprojekten) über Computer-gestützte statistische Verfahren analysieren, um Subtypen von Patienten mit krankheitsassoziierten Veränderungen zu identifizieren und charakterisieren. Der Fokus liegt dabei auf Veränderungen im Mitochondrium, dem Zellorganell, das für die Energieproduktion zuständig ist und in einem Großteil der sporadischen Patienten dysfunktional ist. Das Ziel der bioinformatischen Analysen ist ein genaueres Verständnis der Parkinson-Subtypen mit mitochondrialer Dysfunktion und die Entwicklung neuer diagnostischer Methoden, um diese Subtypen von anderen Parkinson-Untergruppen und Kontrollpersonen zu unterscheiden. Darüber hinaus bietet das Projekt eine Grundlage für die nachfolgende, präklinische Entwicklung therapeutischer Strategien, die spezifisch für die identifizierten Subtypen sind und helfen, das Fortschreiten der Krankheit zu verlangsamen oder Symptome zu lindern.
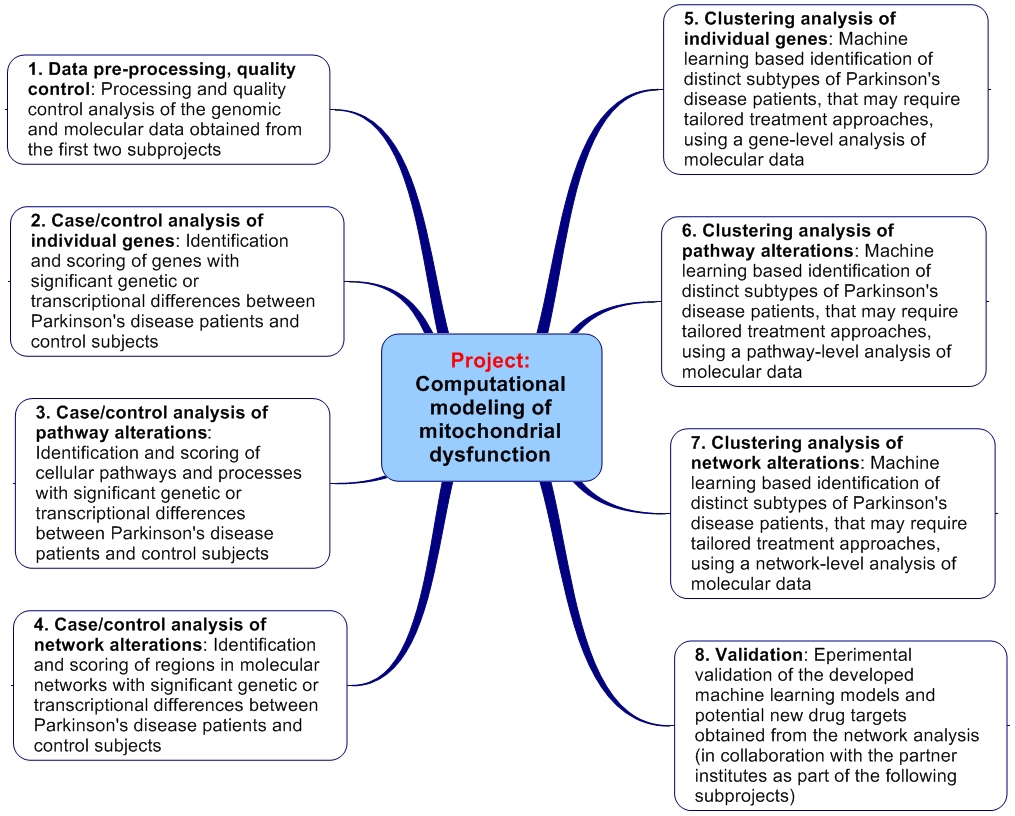
Im ersten Schritt werden über bioinformatische Analysen molekulare und genetische Unterschiede im Mitochondrium zwischen Patienten und Kontrollen, sowie zwischen potentiellen Untergruppen von Patienten, identifiziert und nach ihrer statistischen Signifikanz bewertet (auf der Ebene einzelner Gene/Proteine, der Ebene zellulärer Prozesse, und der Ebene biomolekularer Interaktionsnetzwerke). Dafür nutzen wir existierende Algorithmen, um die Regionen in molekularen Netzwerken mit den stärksten Veränderungen zu bestimmen.
Im zweiten Schritt werden Deregulierungen in den identifizierten Subtpyen für sporadische Parkinson-Patienten mit Veränderungen in Patienten mit einer bekannten genetischen Ursache der Krankheit im Mitochondrium verglichen (d. h. Mutationen in den Genen PINK1 und PARK2). Über maschinelle Lernverfahren sollen dabei Vorhersagemodelle erstellt werden, die Informationen aus der molekularen Aktivität in zellulären Signalpfaden nutzen, um biologische Proben von verschiedenen Parkinson-Subtypen und Kontrollen automatisch zu diagnostizieren. Die Analyse von Veränderungen in der molekularer Netzwerkaktivität wird außerdem Information liefern, um zwischen vorhandenen Therapiestrategien für mitochondriale Dysfunktion eine Auswahl für die Validierung zu treffen und neue alternative Arzneistoffziele vorzuschlagen. In den folgenden Teilprojekten werden schließlich die entwickelten diagnostischen Modelle und Arzneistoffziele experimentell getestet.



