TP 1
Validierung des prädiktiven SYSACT Modells unter organotypischen 3D Bedingungen für TRAIL/IZI1551-Derivate und Trametenib/Dabrafenib
Das auf Kenntnis der apoptotischen Signaltransduktion basierende mathematische Modell (SYSACT) kann das Ansprechen von Melanom-Zelllinien auf Behandlung mit TRAIL und Dakarbazin akkurat vorhersagen. Durch Vereinigung quantitativer Expressionsmuster 17 apoptotischer Regulatoren mit topologischen Informationen der apoptotischen Signaltransduktion wurde eine Informationsbasis geschaffen, die die Ansprechrate prognostizieren kann.
Wir möchten diese Vorhersagen auf komplexere 3D organotypische Hautmodelle übertragen, die Melanom-Sphäroide enthalten, die eine intratumorale Differenzierung analog zu avaskularisierten kutanen Metastasen aufweisen. Sphäroide werden aus Zelllinien generiert, die im SYSACT Modell als sensitiv, wenig responsiv oder resistent gegenüber TRAIL definiert wurden. Diese werden sowohl in vitro als auch integriert in des 3D organotypische Wirtsgewebe analysiert. Die TRAIL Suszeptibilität wird immunhistochemisch, und die Expression definierter Proteine durch standardisierte Lumineszenz-Immundetektion an Paraffinschnitten bestimmt (siehe WP3). Die Ergebnisse werden in das SYSACT Modell integriert, um so die sensitivsten Parameter als prädiktive Marker in einer organotypischen Umgebung identifizieren zu können.
Dabei wird konventionelles TRAIL durch neuartige TRAIL-Rezeptoragonisten ersetzt, die nicht die intrinsische Limitierung therapeutischer Antikörper aufweisen (IZI1551, hexavalente FC-scTRAIL Fusionsproteine; siehe WP4). Dies ermöglicht die Analyse des prädiktiven Potentials von SYSACT, und der therapeutischen Wirksamkeit von Fc-scTRAIL für das Melanom. Vice versa wird die quantitative Expression der 17 in SYSACT identifizierten Apoptose-regulierenden Proteine in Bezug auf die TRAIL Suszeptibilität in Melanomzellen unter 3D Bedingungen getestet.
Parallel werden identische Experimente mit spezifischen BRAF (Dabrafenib)/MEK (Trametinib) Inhibitoren auch in Kombintaion mit Fc-scTRAIL durchgeführt. In Kooperation mit WP2 werden hier BRAFV600E und BRAFwt Melanomzellen mit gekanntem Ras Mutationsstatus in 2D und 3D evaluiert. Hierbei steht besonders die Quantifizierung jener Proteine im Fokus, die bei der Boolschen Modellierung als sensitive Parameter des TRAIL und MEK Signalnetzwerks identifiziert wurden (siehe WP2).
Wir möchten so die Anzahl der Proteine reduzieren, die das Ansprechen auf Kombinationstherapien vorhersagen können, und als Biomarker für die personalisierte Behandlung des Melanoms dienen könnten, zu minimieren.
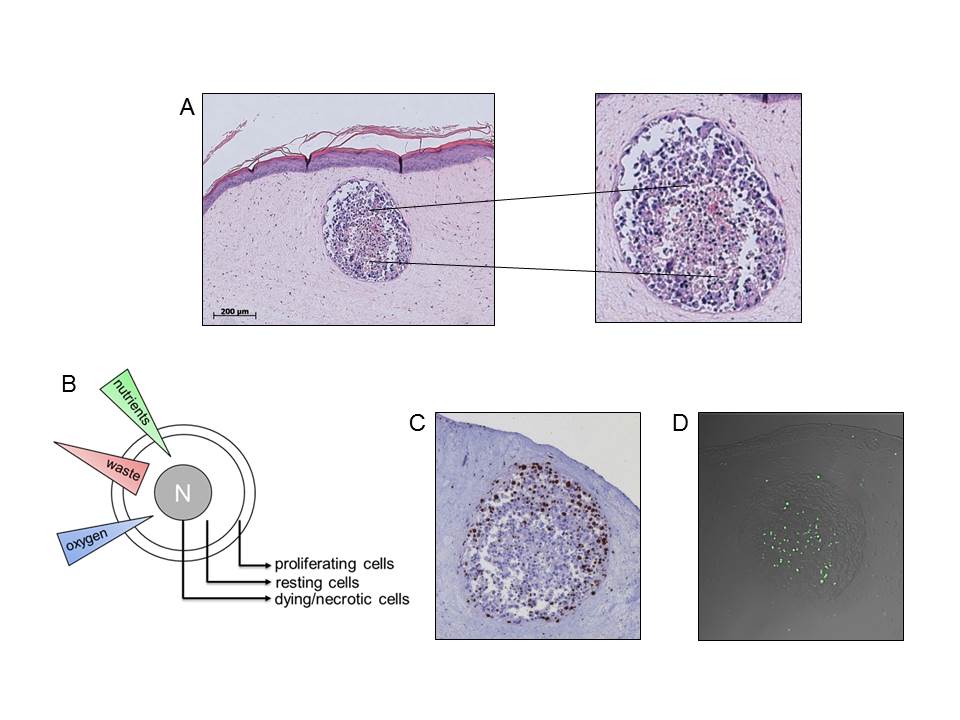
A Sphäroide bestehen aus mindestens 2 Subpopulationen, äußere differenzierte Zellen und pyknotischen Zellen im Zentrum. B Subpopulationen in avaskulären Metastasen entstehen durch ungleiche Verteilung von Nährstoffen, Abfallstoffen und Sauerstoff. C Färbung des im Zellzyklus aktiven Ki-67 Proteins: periphere Zellschichten behalten ihre Proliferationseigenschaften bei. D Färbung von DNA Strangbrüchen mittels der TUNEL Methode: Kernbereich enthält apoptotische Zellen. Vörsmann et al., Cell Death Dis, 2013
Keywords: Melanom, Apoptose, Signaltransduktion, Systembiologie



