TP 6
Mechanistische Multiskalenmodelle
Im Rahmen dieses Teilprojekts wird ein mechanistisches Multiskalenmodell für Herzklappenstenose entwickelt. Das Multiskalenmodell integriert Daten und in Datenanalysen gewonnene Erkenntnisse auf verschiedenen Ebenen (Genom, Hormone, Zelle, Organ, Patient, Population), so dass individuelle Krankheitsverläufe und die Wirksamkeit von Behandlungskonzepten modelliert werden können. Ziel ist es, individuelle Vorhersagen für die Patienten der im Rahmen von SMART durchgeführten klinischen Studie zur Herzklappenstenose zu machen.
Das mechanistische Multiskalenmodell wird beinhalten:
- Physiologie-basiertes Modell des Herz-Kreislauf (CV) Systems
- Modell des Renin-Angiotensin-Aldosteron-Systems (RAAS)
- Barorezeptor Regelkreis-Modell
- Physiologie-basiertes pharmakokinetisches/pharmakodynamisches (PBPK/PD) Modell der pharmakologischen Begleittherapie (Beta-Blocker, Angiotensin-Rezeptorblocker, etc.)
- Zusammenhänge von gemessenen Laborparametern und Pathophysiologie, die in statistischen Analysen aufgedeckt werden.
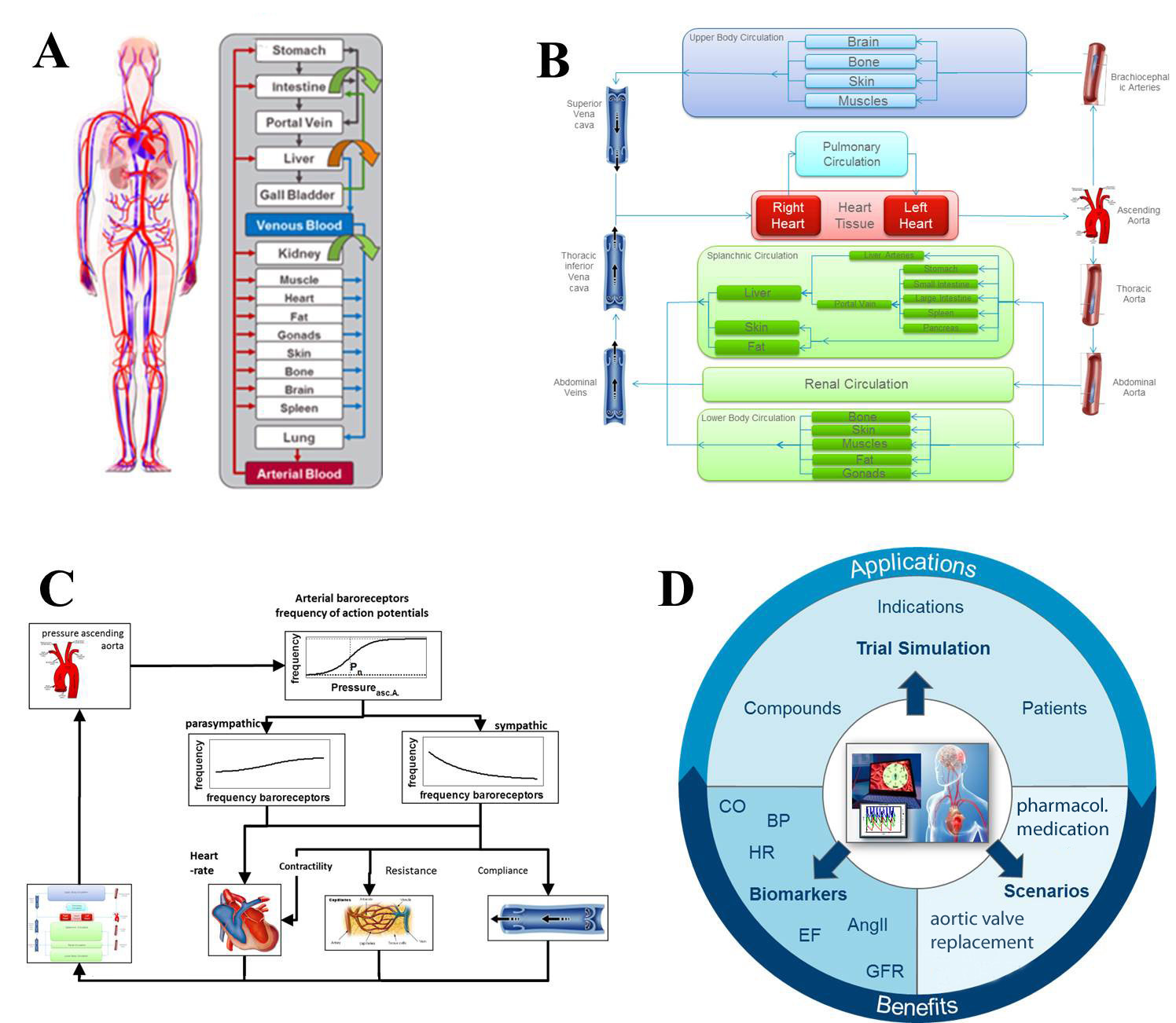
(A) Ausgangspunkt des Herzkreislauf (CV) Modells: die generische PK-Sim Modellstrukur der Systems Biology Software Suite. Links: Anatomie des Blutkreislaufsystems. Rechts: Schematisches Diagramm der Blutflüsse und Organe. Gerundete Pfeile kennzeichnen Exkretionswege
(B) Eine detaillierte Ansicht aller CV Komponenten des Multiskalenmodells. Der systemische Kreislauf besteht aus 4 Gefäßbetten: Oberkörper- (blaue Box), versorgt von den brachiocephalen Arterien, sowie splanchnische, Unterkörper- und renale Zirkulation, die über die abdominale Aorta versorgt werden (grüne Boxen)
(C) In das CV Modell werden weitere Regulationsmechanismen integriert, z.B. der Barorezeptor Reflex Mechanismus, der den Blutdruck über parasympathische und sympathische Effekte auf Herzrate, Kontraktilität, Widerstand und Compliance reguliert.
(D) Das Multiskalenmodell unterstützt klinische relevante Entscheidungen auf unterschiedlichen Ebenen, z.B. durch
• Vorhersage von Veränderungen bestimmter Biomarker (z.B. wie verändert sich der Blutdruck bei Eingriffen in das System?)
• Vorhersage von Szenarien (z.B. wie reagiert der Baroreflex Mechanismus auf einen chirurgischen Herzklappenersatz?)
• Simulation von klinischen Studien.
Diese Modellstruktur erlaubt die Vorhersage relevanter Herz-Kreislaufparameter, wie z.B. Herzrate (HR) oder Cardiac Output (CO) – sogar nach einem chirurgischem Eingriff und unter dem Einfluß von Medikamenten.
Für eine optimierte therapeutische Behandlung ist es notwendig, die Pathophysiologie der Patienten zu verstehen und zu berücksichtigen. Deshalb wird mit statistischen Methoden nach Korrelationen zwischen physiopathologischem Zustand und Auffälligkeiten in „omics-Daten“ gesucht. Hier kommen Algorithmen im Bereich Data Mining zum Einsatz, die im Rahmen des Projekts auch weiterentwickelt werden. Identifizierte Zusammenhänge werden anschließend in die individualisierten Modelle eingebaut, um so eine individuelle Vorhersage zu verbessern.
Zur Anwendung wird das entwickelte Multiskalenmodell entsprechend parametrisiert und an Daten der SMART Projektpartner angepasst. Hierzu werden „State-of-the-art“ Algorithmen aus dem Bereich der Markov-Chain-Monte-Carlo (MCMC) Methodik auf die spezifischen Erfordernisse des Multiskalenmodells angepasst und angewendet. So ist eine detaillierte Analyse von Prädiktivität, Variabilität und Unsicherheiten auf statistisch rigorose Weise möglich.
Das Multiskalenmodell mit den oben aufgeführten Komponenten wird mit der Systems Biology Software Suite von Bayer Technology Services GmbH (www.systems-biology.com) entwickelt. Diese Softwareplattform ist speziell auf Modellierungen im Bereich Life Sciences ausgerichtet.
Keywords: PBPK, cardiovascular modeling, PK-Sim



