TP 2
Bildanalyse zytologischer TMM-Merkmale
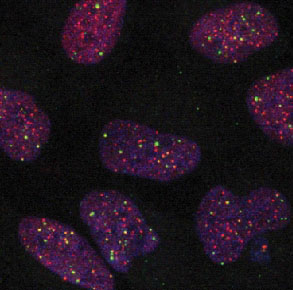
Das Subprojekt SP2 befasst sich mit der Entwicklung und Anwendung von Bildanalyseverfahren zur automatischen Extraktiuon zytologischer Merkmale aus 3D-Mehrkanal-Mikroskopiebildern. Der Fokus liegt auf der Bestimmung von Merkmalen, die für Telomererhaltungsmechanismen ("Telomere Maintenance Mechanisms", TMM) relevant sind. Dazu entwickeln wir ein modellbasiertes Bildanalyseverfahren zur 3D-Lokalisierung und Kolokalisierung subzellulärer Strukturen und wenden es für 3D konfokale Mikroskopiebilder unterschiedlicher Zelllinien (z.B. Glioblastoma) sowie für 3D Mikroskopiebilder von Tissue Microarrays (TMAs) an.

Aus den Bilddaten wird eine Fülle an quantitativen Informationen über zytologische Merkmale unterschiedlicher TMMs extrahiert. Dies beinhaltet beispielsweise die Telomer-Längenverteilung und Heterogenität für die Klassifikation in ALT-positive oder ALT-negative Zellen ("Alternative Lengthening of Telomeres", ALT), die durchschnittliche Telomerlänge sowie die Anzahl und Größe von APBs ("ALT-associated PML Bodies") als Kennzeichen von ALT durch Kolokalisierung von Telomeren und PML-Proteinen. Die automatisch extrahierten zytologischen TMM-Merkmale sind wichtig für die Modellierung von TMM-Netzwerken in Tumoren durch eine integrative systembiologische Herangehensweise sowie zur Stratifikation von Tumorentitäten.
Keywords: Computerbasierte Bildanalyse, zytologische Merkmale, 3D Mehrkanal-Mikroskopiebilder, Telomer-Erhaltungsmechanismus, Alternative Lengthening of Telomeres, 3D Kolokalisierung



