TP 1
Monogenic and oligogenic traits as an entry port to systems medicine
Dieses Projekt setzt auf der Beobachtung auf, dass mittels klassischer Kriterien diagnostizierte chronisch-entzündliche Erkrankungen, insbesondere deren extreme Phänotypen, auch eine signifikante Mendel’sche Krankheitskomponente besitzen, also unter anderem eine Akkumulation von Fällen mit monogen Defekten einschließen. Solche monogenen Komponenten können als Modelle dienen, an denen sich grundlegende molekulare Krankheitsprozesse untersuchen lassen.
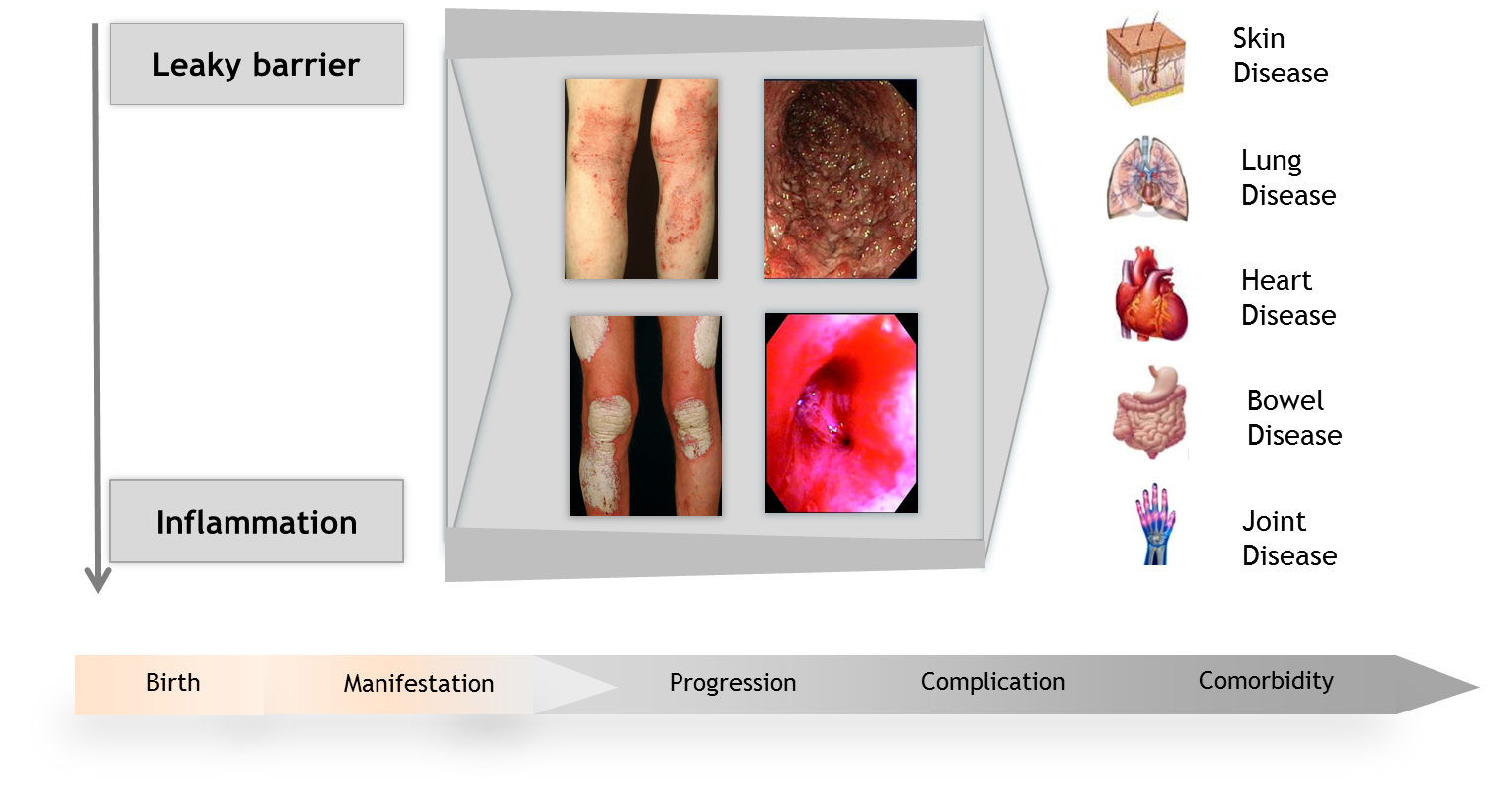
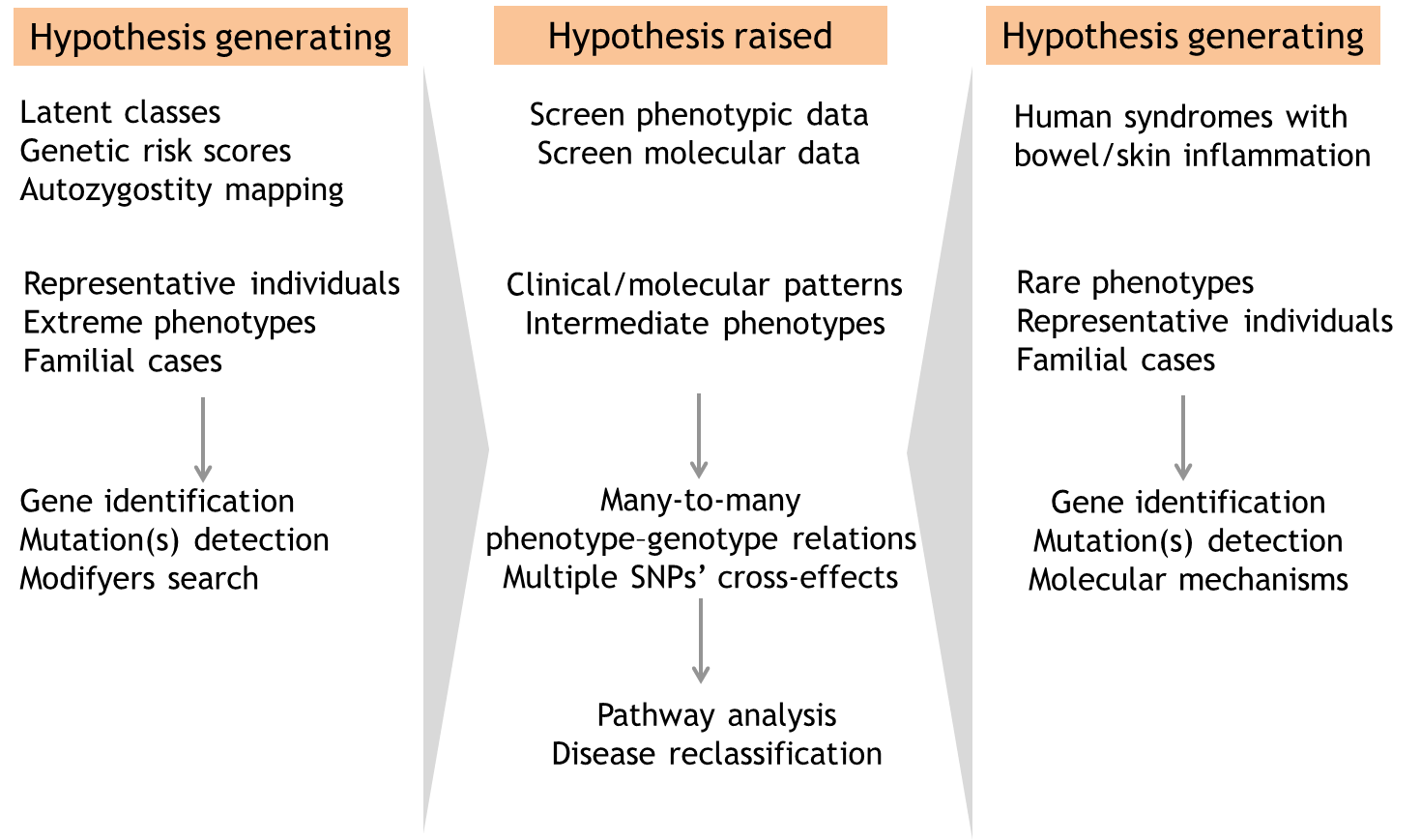
Zudem gibt es Gene, die nur seltene Varianten aufweisen und damit der Entdeckung durch Ansätze aus der komplexen Genetik entgehen können. Solche seltenen Varianten können aber über grosse und kumulative Effkte einen signifikanten Beitrag zur Entstehung von Krankheiten leisten. Die systematische Aufklärung mono- und oligogener Störungen kann so in höherem Maße Einblicke in die Ätiologie und Pathogenese komplexer Krankheiten ermöglichen als dies durch genomweite Assoziationsstudien möglich ist, und zur Entwicklung rational begründeter Krankheitsklassifikationen, Präventiv-und Therapiemaßnahmen beitragen.
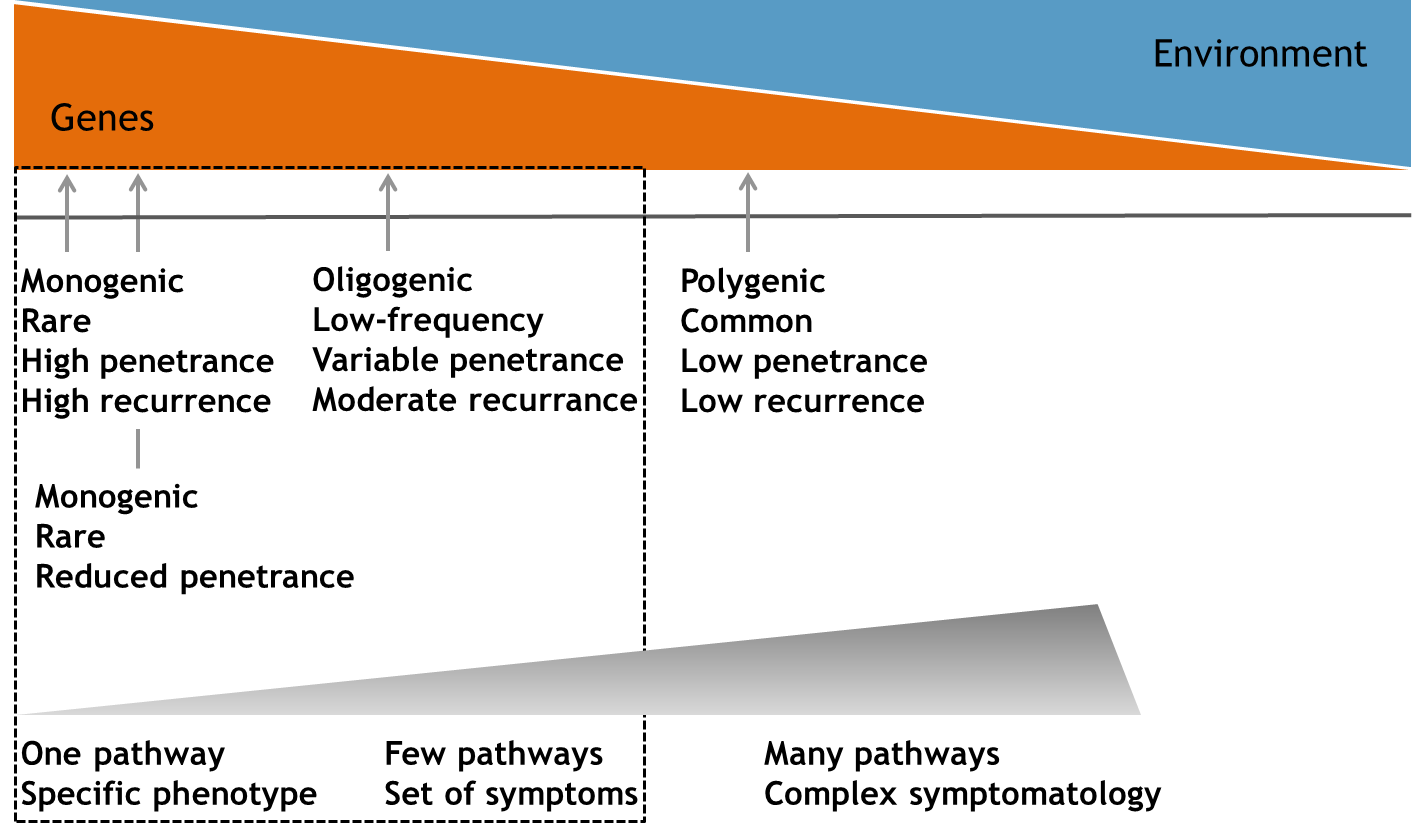
Im Rahmen dieses Projektes sollen daher die Heterogenität von chronisch-entzündlicher Barriere-Erkrankungen durch Stratifikation vorhandener Kollektive anhand klinischer und molekularer Profile reduziert und Biomarker zur Re-Klassifikation der Erkrankungsgruppen generiert werden. Ausreichend homogene Subgruppen von Patientenpopulationen und Familien mit Hinweisen auf das Vorliegen mono- bis oligogener Defekte sollen tiefgehend untersucht werden, um kausale Genvarianten zu identifizieren. Umgekehrt sollen Patienten mit bekannten monogenen Immundefizienzen und begleitender chronisch-entzündlicher Barriereerkrankung tiefgehend charakterisiert und die molekularen Mechanismen einzelner Gendefekte in vitro und im Tiermodell untersucht werden.
Kooperationspartner:
Prof. Dr. med. Michael Kabesch (Regensburg, www.mh-hannover.de/16105.html)
Prof. Bodo Grimbacher (Freiburg, www.zse-freiburg.de/de/CC)
Keywords: Monogenic, oligogenic, complex traits



