TP 3
Identifizierung von Krankheitsassoziierten genregulatorischen Netzwerken
In den letzten 10 Jahren wurde eine Vielzahl genomweiten Assoziationsstudien (GWAS) durchgefuehrt, um die genetische Risikovarianten aller verbreiteter, nicht-uebertragbarer Volkskrankheiten zu finden. Neben der Risikovorhersage fuer Patienten auf Grundlage ihres Genoms und ggf. der Verordnung praeventiver Massnahmen hat sich aus den GWAS vor allem die faszinierende Moeglichkeit ergeben, die molekularen Ursachen dieser Krankheiten zu ergruenden. Um die Risikovarianten zu finden vergleicht man die Genome erkrankter und gesunder Menschen miteinander. Statistische Methode erkennen dann, welche Varianten signifikant mit dem Auftreten der Krankheit assoziiert sind.
Unser eAtheroSysMed-Konsortium untersucht die Ursachen von arteriosklerotischen Krankheiten wie Schlaganfall und koronare Herzkrankheit (KHK), die durch krankhafte Verkalkung der Arterien hervorgerufen werden.
Viele genetische Risikovarianten wurden durch GWAS identifiziert. Leider lassen sich die Mehrzahl der scheinbar von den Varianten betroffenen Gene nicht in die bekannten Entstehungsmechanismen einordnen. In den letzten Jahren steigt die Evidenz, dass der Grund daran liegen koennte, dass die von den Varianten beeinflussten Genen nicht, wie ueblicherweise angenommen, das in der Gensequenz naechstgelegene Gen ist, sondern dass die genetischen Varianten oft die Aktivitaet weiter entfernter Zielgene beeinflussen.
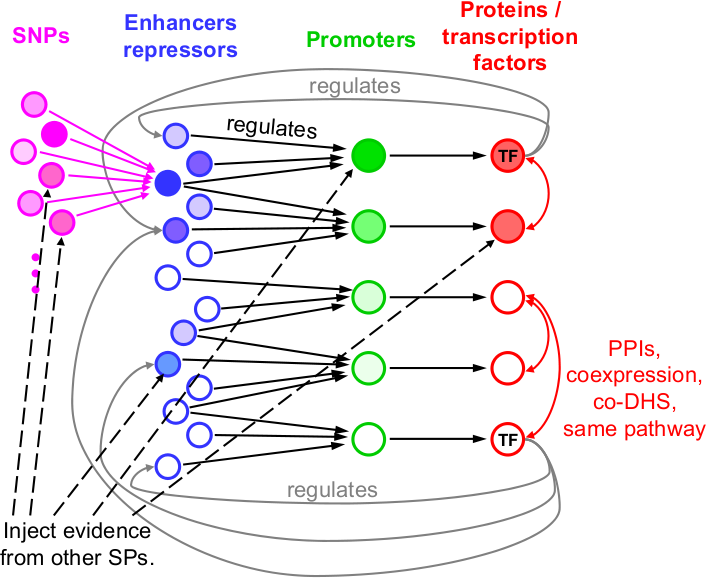
Das erste Ziel unseres Teilprojekt ist daher die Entwicklung statistischer Methoden fuer die Vorhersage von Zielgenen regulativer genetischer Risikovarianten. Unser zweites Ziel ist die Ableitung der genregulatorischen Netzwerke, die durch die Arteriosklerose-assoziierten genetischen Varianten dereguliert werden und so die Entstehung der Krankheit beguenstigen. Hierzu werden wir neuartige statistische und computergestuetzte Methoden der Netzwerkanalyse entwerfen, um genetische Varianten, Krankheitsbild und Genexpressionsdaten zu integrieren mit funktionellen Hochdurchsatz-Daten des menschlichen Genoms. Von diesem Vorgehen versprechen wir uns einen erheblichen Empfindlichkeitsgewinn in der Aufdeckung neuer Krankheitsassoziationen, da unsere Netzwerkanalyse die Evidenz ueber alle genetische Varianten, die eines der Gene eines regulativer Subnetzwerkes beinflussen, aggregiert. Zudem besteht die Hoffnung, dass die deregulierten Netzwerke uns neue therapeutische Behandlungsmoeglichkeiten aufzeigen, indem den deregulierenden Effekten gezielt therapeutisch entgegengewirkt wird.
Keywords: Genome-wide association studies, atherosclerosis, CAD, stroke, computational biology, systems medicine, network analysis, enhancers, regulatory SNPs, regulatory networks



