TP 4
SNP-vermittelte miRNA (Dys)-Regulation bei Atherosklerose
Die Rolle von microRNAs (kleinen, nicht-kodierenden RNAs) bei der Entstehung von Atherosklerose ist noch weitgehend unklar, doch gibt es Hinweise, dass genetische Varianz (single nucleotide polymorphisms, SNPs) die Expression oder Aktivität von microRNAs ungünstig beeinflussen und so atherosklerotische Prozesse in Gang setzen kann. Im Teilprojekt 4 verfolgen wir in sieben Arbeitspaketen (WPs) zwei Hypothesen: 1) SNPs in microRNA-Genen oder anderen Genen (z.B. von Transkriptionsfaktoren) verändern die Expression von microRNAs und 2) SNPs in microRNA-Bindestellen beeinträchtigen die Regulierung bestimmter mRNAs durch diese microRNAs (Abb.).
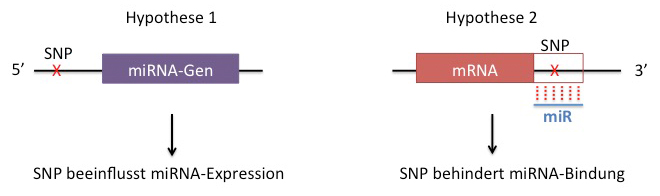
Ziel unserer Forschung ist es, solche genetischen Varianzloci umfassend zu identifizieren und ihre Auswirkung funktionell zu beschreiben. Hierzu quantifizieren wir die Expression von je 1.700 miRNAs in Proben von 150 Patienten mit koronarer Herzkrankheit (KHK) oder Schlaganfallhistorie und vergleichen diese Daten mit Genotypdaten der Patienten (WP4.1). Hierdurch werden SNP-enthaltende Loci identifiziert, die die Expression von micro-RNAs beeinflussen (sog. eQTLs). Im nächsten Schritt werden neue und auch bereits bekannte eQTL mit dem Auftreten von KHK/Schlaganfall im Patientenkollektiv korreliert (WP4.2). Nun wird für jene microRNAs, die im Zusammenhang mit eQTL stehen, experimentell untersucht, ob diese die Proliferation von Gefäßmuskelzellen (VSMCs) - einem Merkmal der Atherosklerose - steuern. Hierzu wird durch Transfektion von VSMCs mit sog. “miR-Mimics” oder Inhibitoren (antimiRs) die zelluläre Konzentration oder die Aktivität der microRNAs manipuliert (WP4.3). Am Ende der Arbeiten zu Hypothese 1 sollten genetische Varianzen bekannt geworden sein, die die Expression von microRNAs verändern und Atherosklerose entstehen lassen. Zur Hypothese 2 werden Daten von KHK-/Schlaganfall-Assoziationsstudien (GWAS) auf SNPs in microRNA-Bindestellen untersucht, um Kandidatengene zu identifizieren (WP4.4). Wie sich diese SNPs auf die Fähigkeit von microRNAs, ihre Ziel-mRNAs zu regulieren, auswirkt, validieren wir anhand von Luciferase-Reporterassays (WP4.5). Die Frage, welche Signalwege von den mit Atherosklerose assoziierten microRNA-SNPs betroffen sind, werden wir in silico durch “cross-OMICS enrichment” untersuchen (WP4.6). Als Validierung unserer Ergebnisse in vivo wollen wir zwei SNP-“belastete” Zielgene von microRNAs gentisch deletieren und in vivo in Atherosklerose-Szenarien untersuchen (WP4.7).
Keywords: SNP, microRNA, eQTL



