TP 1 - CancerTelSys
TMM-Marker des Genoms, Epigenoms und Transkriptoms
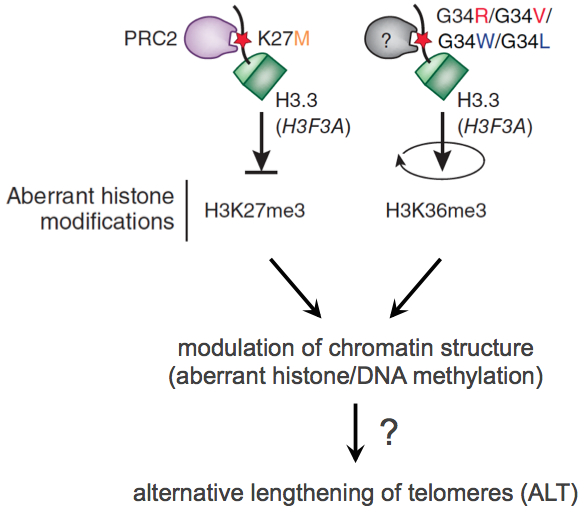
In Subprojekt SP1 erarbeiten wir eine erste Zusammenstellung von Merkmalen, die für aktive Telomererhaltungsmechanismen ("Telomere Maintenance Mechanism", TMM) relevant sind. Bereits erhobene Genexpressions-, DNA-Sequenz- und Methylomdaten werden ausgewertet, um Schlüsseleigenschaften der Telomerverlängerungsmechanismen in unterschiedlichen Tumorproben zu identifizieren. Gestützt auf die Analyse dieser Merkmale in Prostata- und Glioblastom-Tumorgewebeproben erstellen wir eine Liste von identifizierten deregulierten (epi)genomischen TMM-Merkmalen sowie eine Liste von RNAs, die für aktive TMMs charakteristisch sind. Diese Listen werden in die TelNet Datenbank integriert, die ebenfalls Gene beinhaltet, die für die Telomerbiologie relevant sind. Die TelNet-Datenbank ermöglicht eine systematische Evaluation der TM-Merkmale, ihre Integration in ein TM-Signalwegmodell und genomweite Mutationsanalysen von Krebsgenomen. Zusätzlich bringen wir die Entwicklung von Standard-Protokollen für die Analyse der Hochdurchsatz-Sequenzierdaten, insbesondere in Bezug auf nicht-kodierende RNAs, voran. Je nach Vorhandensein von alternativen Telomerverlängerungsmechanismen ("Alternative Lengthening of Telomeres", ALT) sind mehrere hundert vorhandene Tumorproben bereits bezüglich ihrer Telomerlängen-Heterogenität in ALT-positive und ALT-negative Tumoren eingestuft worden, wodurch eine erste grobe Gruppierung sofort erstellt werden kann. Das Subprojekt SP1 konzentriert sich hierbei auf die Analyse der Komponenten ATRX/DAXX/H3.3 in kindlichen Glioblastomen (GBM) und Riesenzelltumoren (GCTB) im Hinblick auf ihre Verbindung zur ALT in diesen Tumorentitäten. Diese Gene wiesen in Vorarbeiten der Arbeitsgruppen Pfister, Plass und Sauter sogenannte „Driver“-Mutationen auf, welche die Entstehung und das Wachstum von GBM und Prostatakrebs besonders stark begünstigen. Diese Mutationen gehen mit drastischen Änderungen des globalen DNA-Methylierungsmusters einher, welche ebenfalls als ein Parameter in die TMM-Analyse mit einbezogen werden. Weiterhin von besonderem Interesse sind bestimmte, mit dem aktiven TMM assoziierte Reparaturproteine.